Double Selection
Esse é um post de um tema bem importante que eu não vejo muita gente dando atenção - de repente é ignorância minha. O problema é bem simples: você vai estimar um efeito de tratamento. Você tem uma infinidade de controles. Você decide selecionar os controles usando algum método.
Isso gera uma distribuição bimodal do parâmetro de tratamento se a variável excluída afeta o tratamento.
Eu não sei se posto dessa maneira é extremamente surpreendente: soa como viés de variável omitida.
Para mostrar o efeito disso, eu vou fazer uma simulação. Vai ter uma variável \(x\) e uma variável \(trat\). No fim a gente vai querer saber o efeito do tratamento sobre a variável \(y\), que é afetado por x e pelo trtamento. A variável \(trat\) vai ser influenciada por x, e eu vou fazer a regressão \(y \sim x + trat\), mas vou excluir \(x\) quando o p-valor for maior que 5%. Eu podia usar alguma coisa mais moderna, tipo LASSO, mas o problema é de seleção de variável, então qualquer maneira de selecionar as variáveis vai gerar o problema. Eu vou colocar 100 observações, para maximizar a chance da gente excluir x.
Eu vou replicar isso umas 2000 vezes e vamos olhar o histograma do coeficiente de tratamento:
set.seed(2126111)
cofs <- rep(NA,2000)
dropped <- rep(NA,2000)
for(i in 1:2000){
x <- rnorm(100)
trat <- 2*x + rnorm(100)
trat <- trat > 0
y <- 1.5*trat + 0.5*x + rnorm(100,sd=2)
reg <- lm(y ~ x + trat)
p_val <- summary(reg)$coefficients[2,4]
if(p_val < 0.05){
cofs[i] <- coef(reg)[3]
dropped[i] <- 0
} else {
reg <- lm(y ~ trat)
cofs[i] <- coef(reg)[2]
dropped[i] <- 1
}
}Tem bastante coisa acontecendo nas linhas acima, então vamos passar as coisas com calma: primeiro, 0 tratamento começa como uma variável contínua que depende de x e de um erro com variância 2. Na linha seguinte, eu transformo essa variável em uma coisa binária: se for maior que 0, o sujeito é tratado. O problema seguinte é o p-valor de x na regressão para avaliar o efeito de tratamento: se for abaixo de 0.05, a regressão segue inalterada. Senão, ai a gente faz só a regressão de y no tratamento. O efeito real do tratamento é 1.5. Veja que eu tenho uma variável que diz quantas vezes a variável x foi excluida. Na simulação acima isso ocorreu em 1189 casos. Vamos ver o histograma do efeito de tratamento:
cofs_df <- data.frame(cofs = cofs)
ggplot(cofs_df,aes(cofs)) + geom_histogram()## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.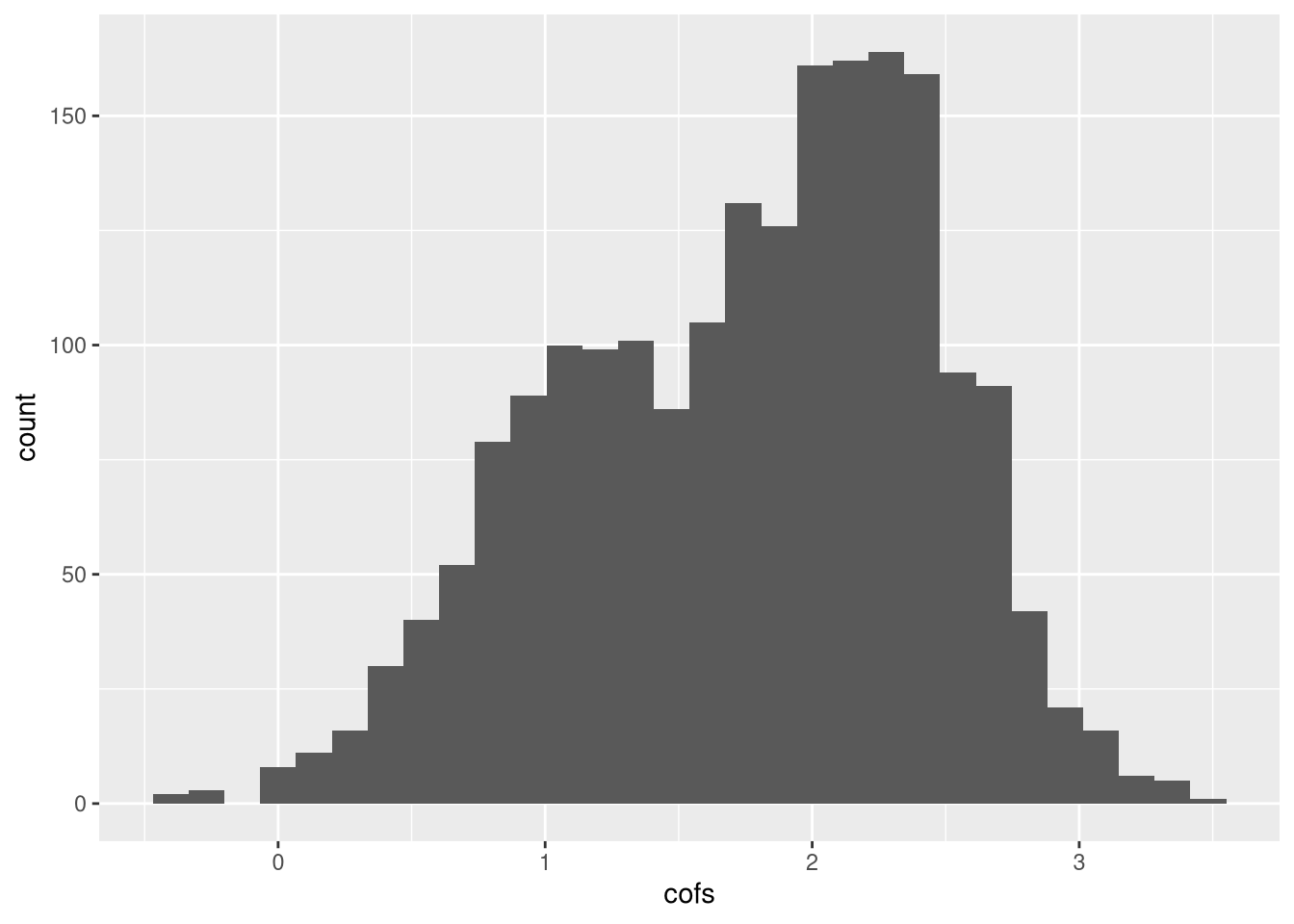
Essa distribuição é bastante esquisita. Como isso afeta os testes de hipótese?
p_vals <- rep(NA,2000)
dropped <- rep(NA,2000)
for(i in 1:2000){
x <- rnorm(100)
trat <- 2*x + rnorm(100)
trat <- trat > 0
y <- 0.5*x + rnorm(100,sd=2)
reg <- lm(y ~ x + trat)
p_val <- summary(reg)$coefficients[2,4]
if(p_val < 0.05){
p_vals[i] <- summary(reg)$coefficients[3,4]
dropped[i] <- 0
} else {
reg <- lm(y ~ trat)
p_vals[i] <- summary(reg)$coefficients[2,4]
dropped[i] <- 1
}
}
pp <- mean(p_vals < 0.05)Nós rejeitamos a hipótese nula quando ela é verdadeira em 28.4% dos casos, quando nós deveríamos rejeitar para apenas 5% dos casos.
Como contornar isso? Bom, você sempre pode encher de todos os controles possíveis e imagináveis. É claro que podemos ter um caso em que o número de controles é maior que o número de observações, isso não funciona. É ai que entra uma ideia simples e super esperta: no lugar de você olhar só se a variável de controle só na regressão de y no tratamento, também olhe se o controle é significativo na regressão do tratamento. Esse procedimento é chamado de double selection. O procedimento anterior que eu fiz é chamado de single selection. Vamos testar isso:
cofs <- rep(NA,2000)
dropped <- rep(NA,2000)
for(i in 1:2000){
x <- rnorm(100)
trat <- 2*x + rnorm(100)
trat <- trat > 0
y <- 1.5*trat + 0.5*x + rnorm(100,sd=2)
reg <- lm(y ~ x + trat)
p_val <- summary(reg)$coefficients[2,4]
reg2 <- lm(trat ~ x)
p_val2 <- summary(reg2)$coefficients[2,4]
if(p_val < 0.05 || p_val2 < 0.05){
cofs[i] <- coef(reg)[3]
dropped[i] <- 0
} else {
reg <- lm(y ~ trat)
cofs[i] <- coef(reg)[2]
dropped[i] <- 1
}
}cofs_ds_df <- data.frame(cofs = cofs)
ggplot(cofs_ds_df,aes(cofs)) + geom_histogram()## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.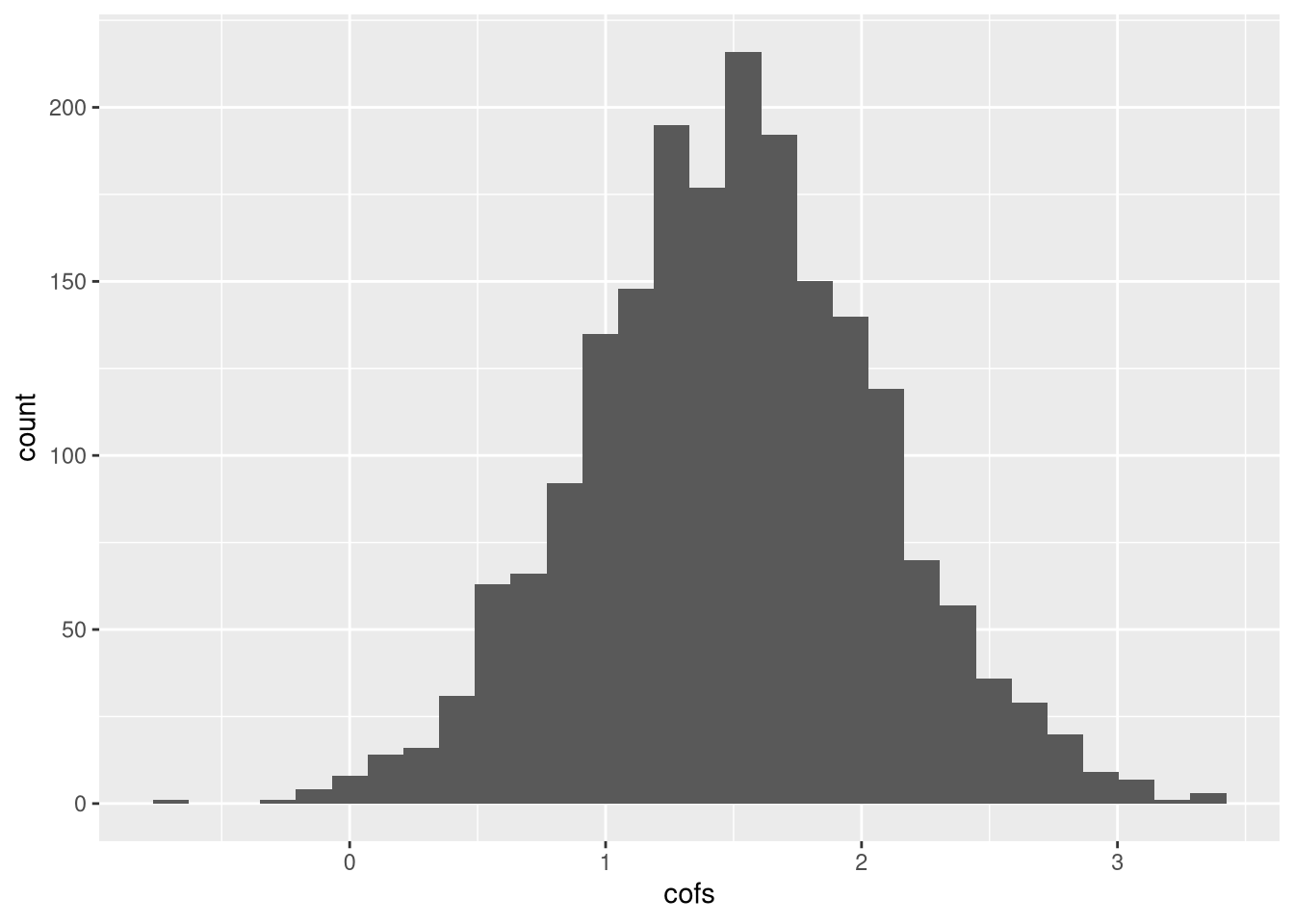
A distribuição fica bem melhor. Vamos ver como fica o erro de tipo I a 5% quando o tratamento não tem efeito:
p_vals <- rep(NA,2000)
dropped <- rep(NA,2000)
for(i in 1:2000){
x <- rnorm(100)
trat <- 2*x + rnorm(100)
trat <- trat > 0
y <- 0.5*x + rnorm(100,sd=2)
reg <- lm(y ~ x + trat)
p_val <- summary(reg)$coefficients[2,4]
reg2 <- lm(trat ~ x)
p_val2 <- summary(reg2)$coefficients[2,4]
if(p_val < 0.05 || p_val2 < 0.05){
p_vals[i] <- summary(reg)$coefficients[3,4]
dropped[i] <- 0
} else {
reg <- lm(y ~ trat)
p_vals[i] <- summary(reg)$coefficients[2,4]
dropped[i] <- 1
}
}
pp <- mean(p_vals < 0.05)Nós rejeitamos a hipótese nula quando ela é verdadeira em 4.65% dos casos, muito mais próximo do ideal.
Bibliografia
O Gabriel Vasconcelos fez um post muito bom no blog dele. Está em inglês. (Gabriel já “apareceu” aqui por ser autor de um pacote que usa critério de informação no LASSO).
O artigo que gerou double selection é do Victor Chernozhukov, Alexandre Belloni e Christian Hensen, no arxiv