Alguns pequenos problemas de clustering k-means
No meu último post mostrei como podíamos usar clustering \(k\)-means para tentar identificar - com relativo sucesso - cursos de medicina no ProUni. Hoje, ao contrário de mostrar um uso interessante de \(k\)-means, quero mostrar um problema do algoritimo relacionado a uma de suas hipoteses.
Hipoteses são ferramentas curiosas. Quem é familiarizado com economia sabe como a profissão as ama. Num geral, elas funcionam como foram concebidas: maneiras de tirar ruído e complexidade de uma questão que não são particularmente relevantes aqui. Uma das rules of thumb da profissão para modelagem é a de que hipoteses são simplificadoras apenas na medida em que não alteram as conclusões principais do modelo.
Pois, supor informação (quasi-)perfeita é absolutamente razoável na maioria dos mercados. Existe alguma assimetria relevante de inforção entre feirante e comprador de bananas? Entre concessionária e comprador de carro? Supor algum tipo de comportamento maximizador de lucro (ou de utilidade) também soa um tanto quanto absurdo, mas veja bem, funciona. Quando capital fica relativamente ao trabalho mais barato, firmas automatizam. Quando tomates ficam mais caros, consumidores compram menos. Quase como se maximizadores racionais caminhassem sobre a terra.
Mas e \(k\)-means?
Vamos lembrar brevemente da matemática por trás do clustering \(k\)-means, a função objetivo do procedimento (não confundir com o algoritimo para computar o problema em si). \(k\) é o número de clusters, \(S_i\) é conjunto de elementos do i-ésimo cluster, \(\mu_i\) é a média do i-ésimo cluster.
\[ \text{arg min}_S \sum_{i=1}^k \sum_{x_j \in S_i} || x_j - \mu_i ||^2 \]
O único instrumento desse problema de otimização é \(S\), \(k\) é um parâmetro dado. Pois, ao escolher um \(k\) em especifico, estamos supondo que existem \(k\) agrupamentos nos dados. E se não for bem assim? Vamos a um exemplo:
##### Começaremos gerando dados aleatórios
##### Seja n o tamanho da amostra, o leitor pode alterar se quiser
n = 100000
#### Geraremos um vetor aleatório no R^2
x <- rnorm(mean = 0,
sd = 1,
n= n) ##média 0 e variância unitária nos dá uma normal padrão
y <- rnorm(mean = 0,
sd = 1,
n = n)
amostra1 <- data.frame(x, y)library(ggplot2)
library(dplyr)
amostra1 %>%
ggplot(aes(x=x, y=y))+
geom_point(color = "dark blue")+
geom_density_2d(color = "light blue")+
geom_vline(aes(xintercept=mean(x)),
color="black", linetype="dashed", size=1)+
geom_hline(aes(yintercept=mean(y)),
color="black", linetype="dashed", size=1)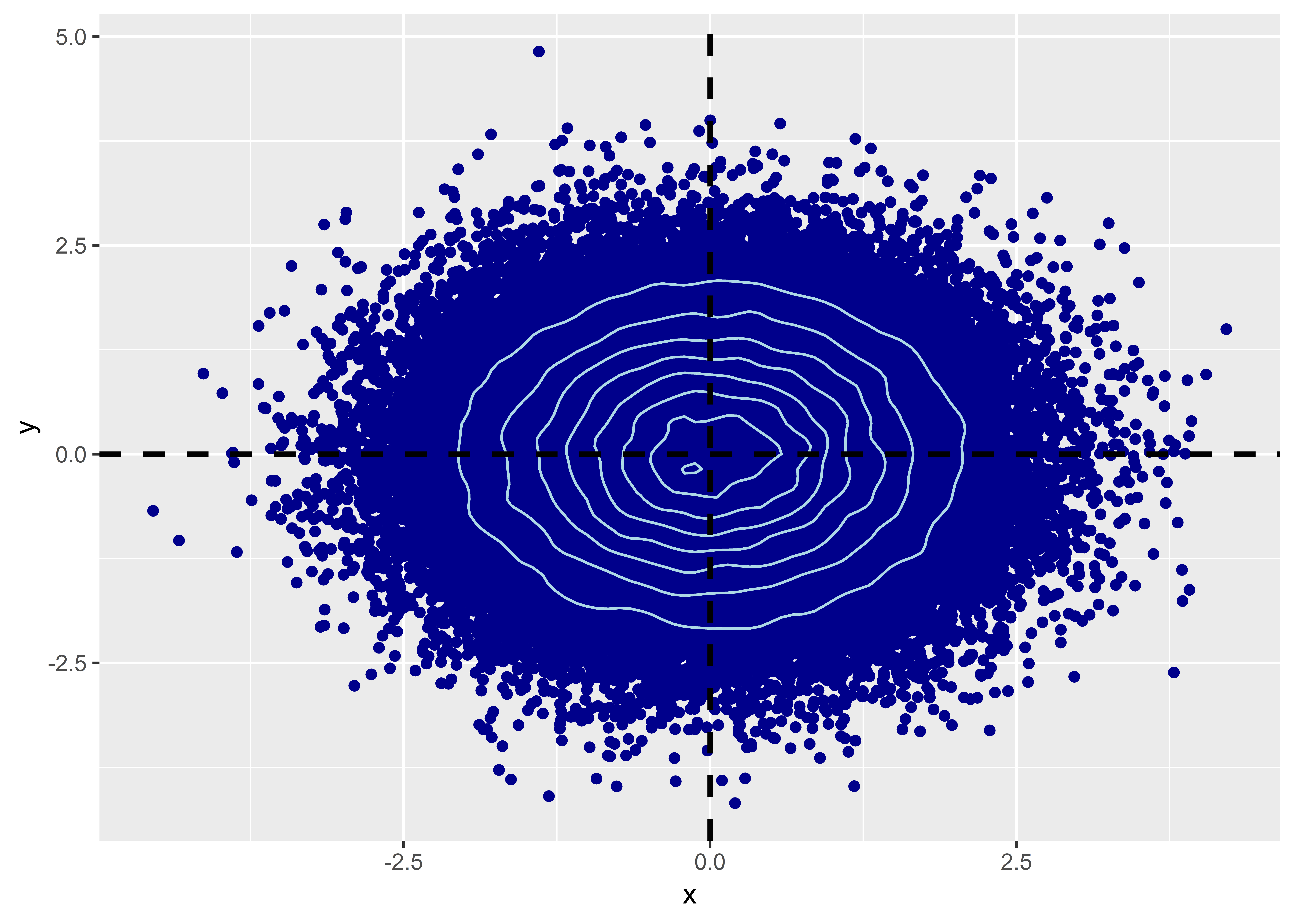
Existe claramente só um agrupamento, mas podemos detectar quantos agrupamentos quisermos ao definir um \(k\) desejado. Antes, vamos avaliar qual seria o \(k\) ótimo segundo o Método do Cotovelo:
wssplot <- function(data, nc=20, seed=1234){
wss <- (nrow(data)-1)*sum(apply(data,2,var))
for (i in 2:nc){
set.seed(seed)
wss[i] <- sum(kmeans(data, centers=i)$withinss)}
plot(1:nc, wss, type="b", xlab="k",
ylab="WGSS")}
wssplot(amostra1)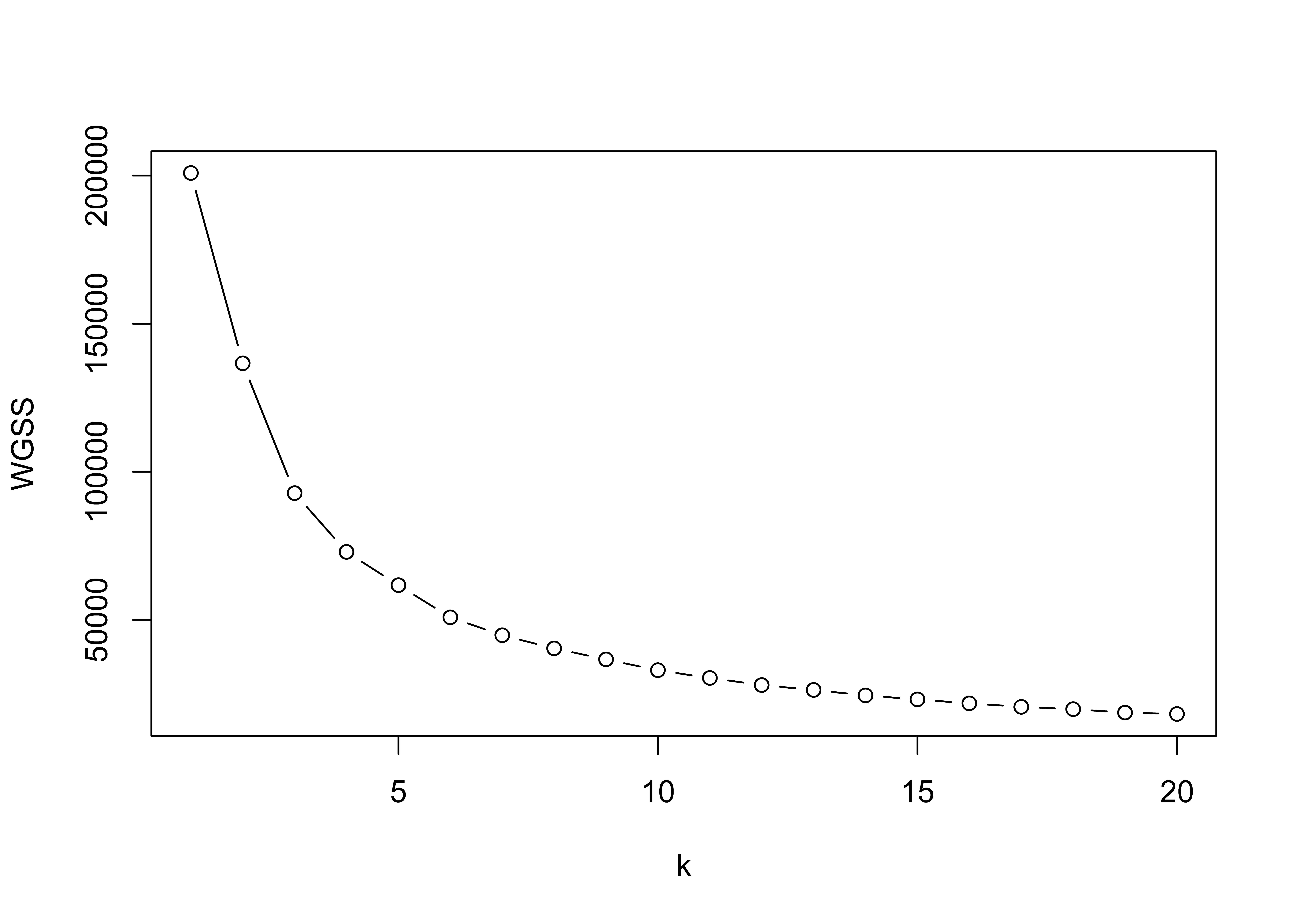
\(k=6\) parece ser compatível com um ponto em que adicionar agrupamentos não rende uma queda substancial no WGSS, então vamos tentar esse número e ver o que acontece.
kmeans_amostra1 <- kmeans(amostra1,
centers = 6)
amostra1 %>%
ggplot(aes(x=x, y=y))+
geom_point(color = kmeans_amostra1$cluster)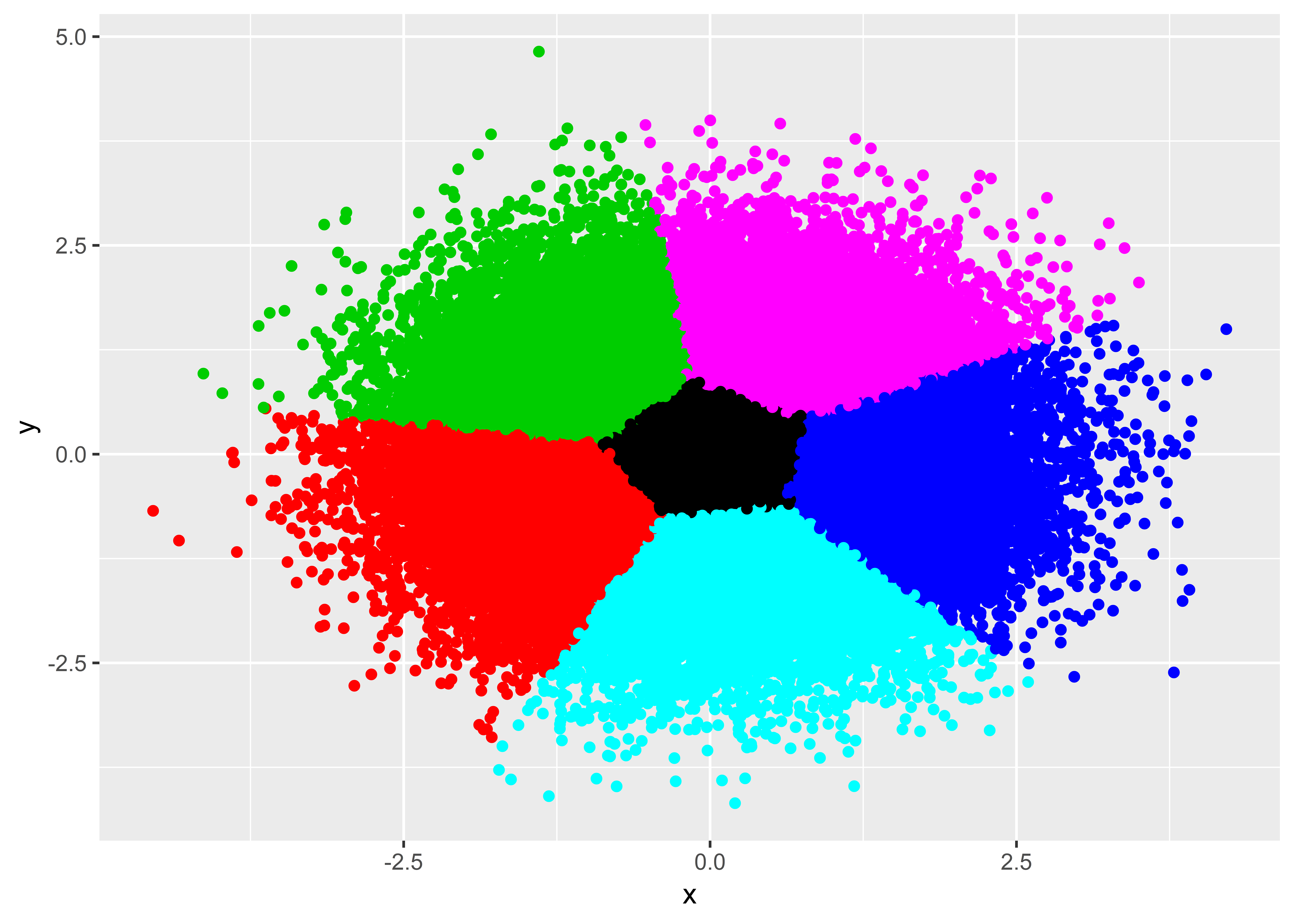
Inclusive, podemos formar padrões similares simplesmente aumentando \(k\) e particionando dados homogêneos em agrupamentos menores - apesar de não existirem de fato.
kmeans_amostra1_2 <- kmeans(amostra1,
centers = 10)
amostra1 %>%
ggplot(aes(x=x, y=y))+
geom_point(color = kmeans_amostra1_2$cluster)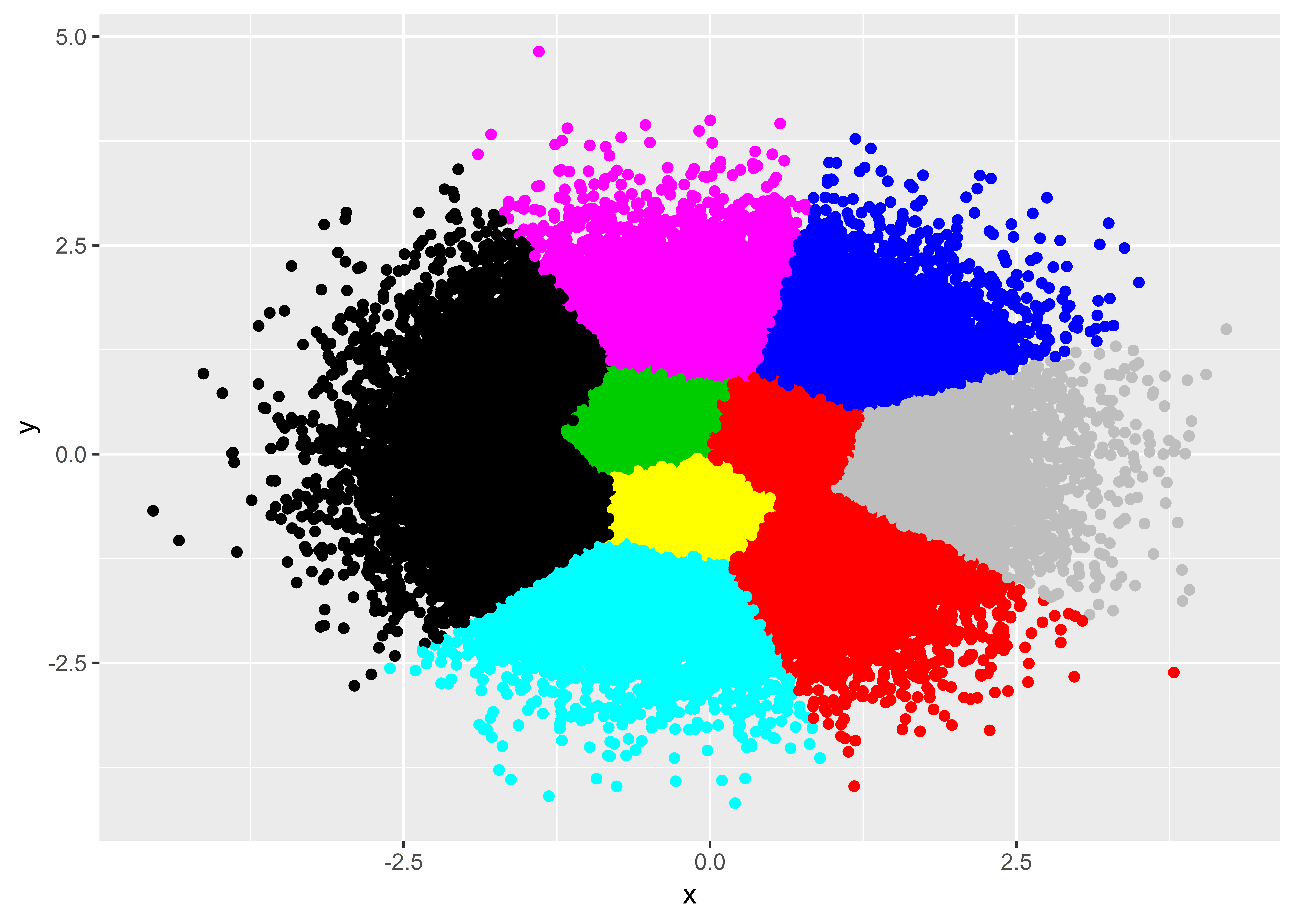
kmeans_amostra1_3 <- kmeans(amostra1,
centers = 50)
amostra1 %>%
ggplot(aes(x=x, y=y))+
geom_point(color = kmeans_amostra1_3$cluster)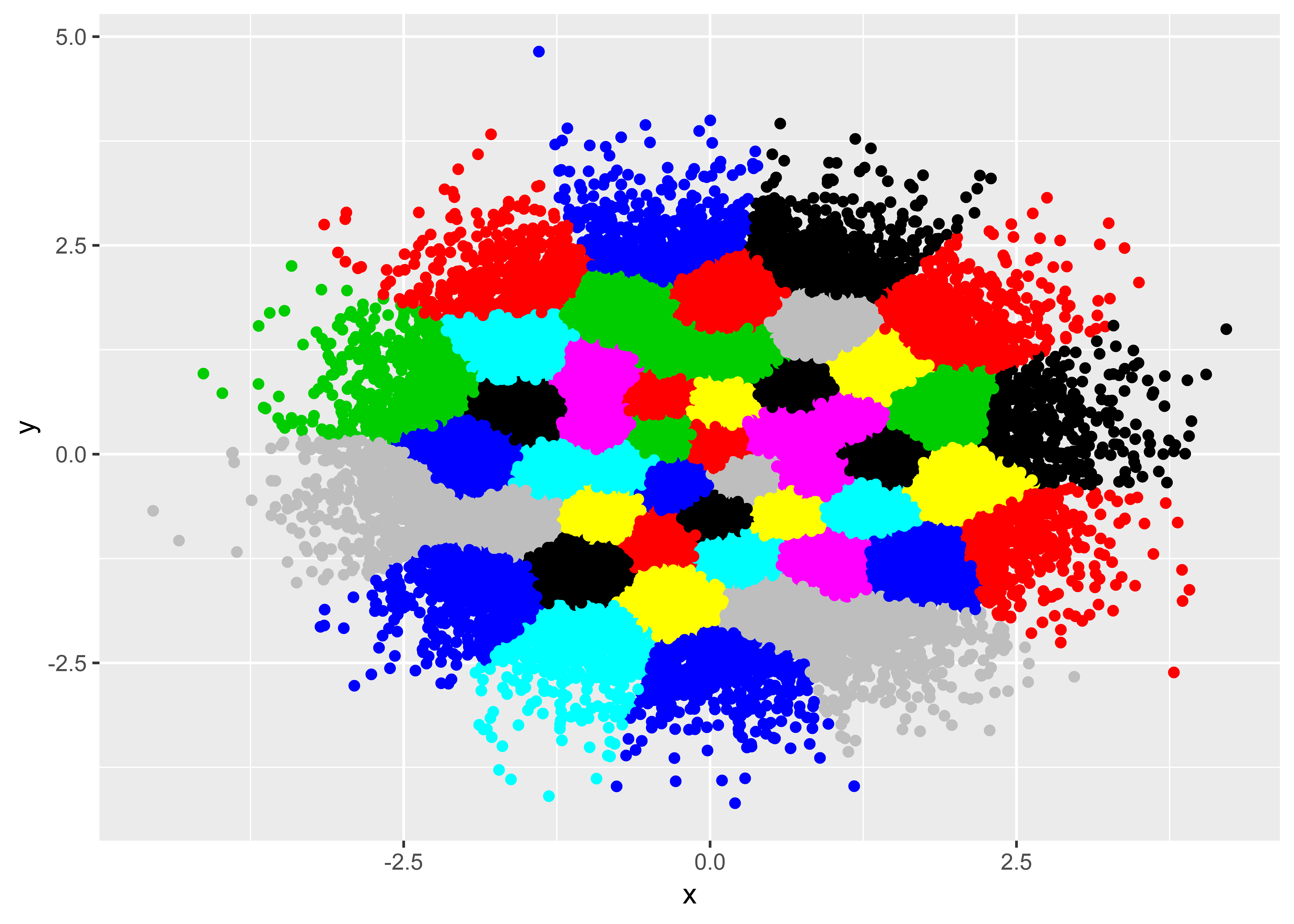
Observem que WGSS em função de \(k\) tem comportamento assintótico bem claro, embora não necessariamente monotônico: converge a zero.
Mas e se existirem \(k\) agrupamentos?
Aí, meu caro leitor, estamos conversando. Vamos gerar novos dados, agora com agrupamentos separados.
k = 8 ## numero de agrupamentos
m = 100 ## tamanho da amostra em cada agrupamento
sd = .2 ## .2 gera identificação limpa dos agrupamentos em alguns casos
datalist = list() ## Lista para salvar os DFs com cada cluster
for (i in 1:k){
x <- rnorm(mean = i,
sd = sd,
n=m)
y <- rnorm(mean = (k-i),
sd = sd,
n=m)
datalist[[i]] <- data.frame(x,y)
}
amostra2 <- do.call(rbind, datalist) ## empilhamos os clusters
amostra2 %>%
ggplot(aes(x=x, y=y))+
geom_point(color = "blue")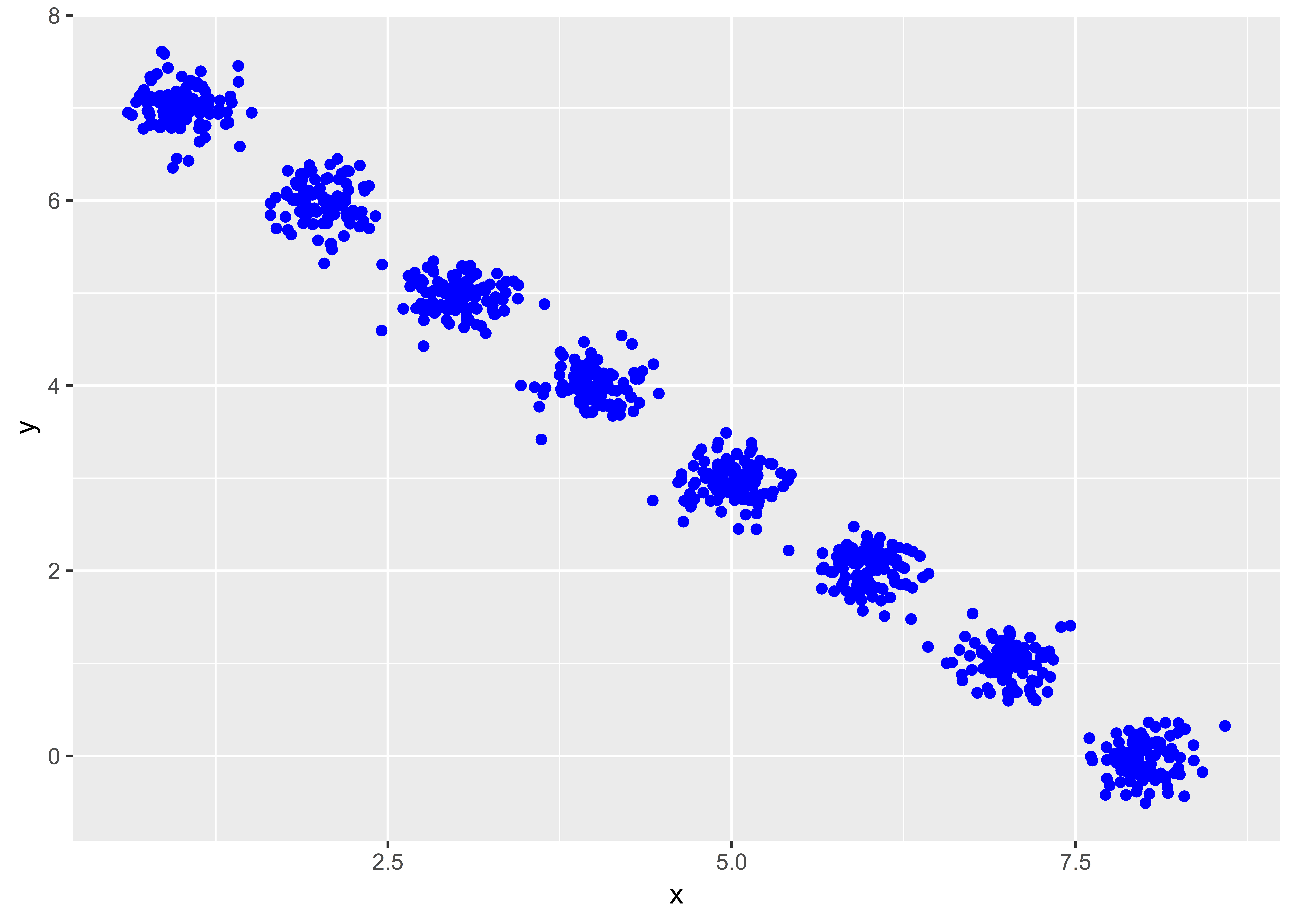
wssplot(amostra2, nc = 10)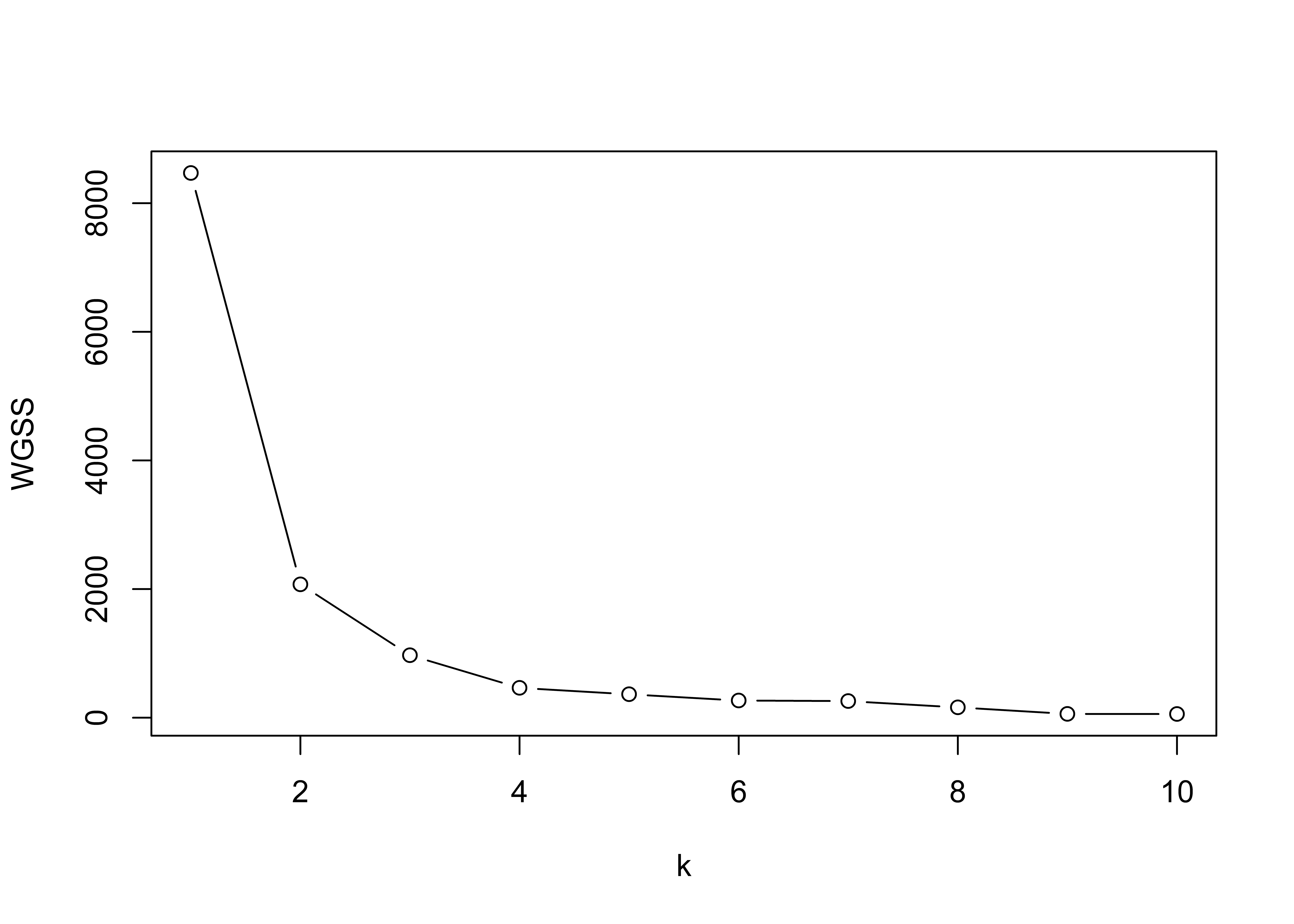 Um pequeno exercício: em dados claramento agrupados em 8 núcleos, qual a diferença do WGSS quando temos \(k=8\), fiel aos dados, e \(k=10\), um exagero?
Um pequeno exercício: em dados claramento agrupados em 8 núcleos, qual a diferença do WGSS quando temos \(k=8\), fiel aos dados, e \(k=10\), um exagero?
teste8 <- kmeans(amostra2,
centers = 8)
teste10 <- kmeans(amostra2,
centers = 10)
teste8$tot.withinss / teste10$tot.withinss #dividindo o WGSS de um pelo outro## [1] 0.4025824Aqui entra de novo o componente estocástico. Já consegui 3,5% maior, já consegui 40% menor. Quanto você, leitor, achou?
kmeans_amostra2 <- kmeans(amostra2,
centers = k)
amostra2 %>%
ggplot(aes(x=x, y=y))+
geom_point(color = kmeans_amostra2$cluster)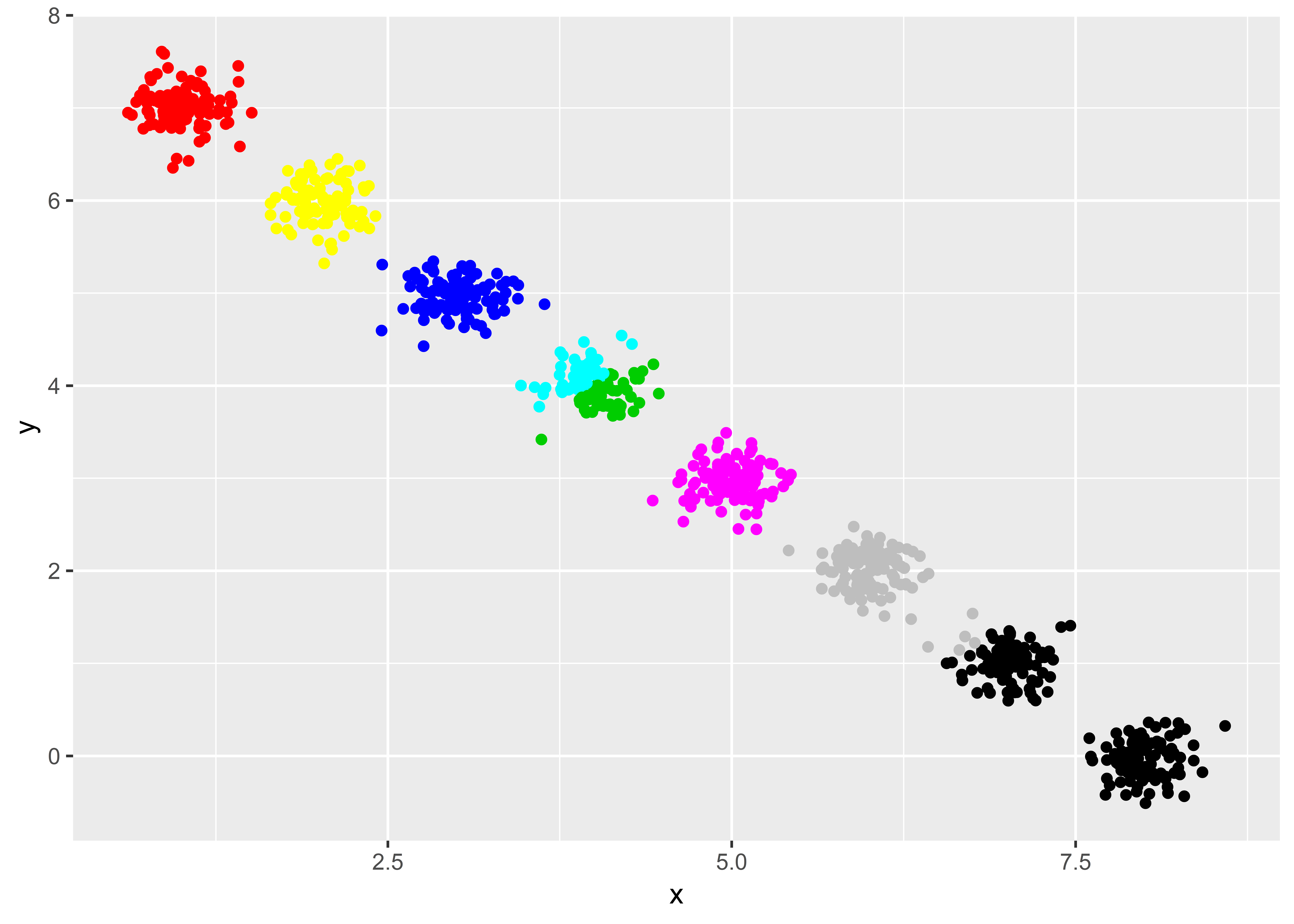
Você talvez precise gerar algumas amostras antes de conseguir uma identificação limpa de cada agrupamento.
Acho que é isso
Queria mostrar brevemente as limitações de (i) uma hipotese através dessa ilustração interessante e (ii) do Método do Cotovelo, computacionalmente simples, mas dependente de interpretação.
(Como sempre, você pode reproduzir isso tudo com esse script)