O LASSO
Este post vai tratar de um método de machine learning muito interessante e relativamente simples: o LASSO. LASSO significa Least Absolute Shrinkage and Select Operator. Como o nome sugere, o LASSO seleciona quais regressores são relevantes e quais não são. Ou seja, suponha que você é um pesquisador que tem 50 variáveis que são possíveis candidatos a variáveis explicativas de uma variável de interesse. O LASSO permite que você dê os 50 regressores para o computador e ele escolha quais são os relevantes. O LASSO está centrado na ideia de esparsidade: poucos coeficientes vão ser diferentes de zero; poucas variáveis vão ser relevantes numa regressão.
Uma pergunta justa é: por que não simplesmente testamos isso “na mão”? Isso é, fazemos uma regressão com todas as variáveis, uma sem a primeira variável da base de dados, uma outra sem a segunda… e no fim vemos qual a melhor regressão? Além de problemas de teste - lembre que todo teste tem uma chance de você estar tomando a decisão errada - isso seria um inferno: com relés 50 variáveis, precisaríamos de regressões, ou seja, 1.1258999^{15} regressões: esse é um número com 15 zeros.
A ideia original vem de um paper de 1996 de Robert Tibshirani. Não temos solução fechada para encontrar o estimador de LASSO, ao contrário do MQO, onde podemos expressar o estimador como uma multiplicação de matrizes. Portanto, é um estimador que seria impossível sem um computador. Mas a ideia é incrivelmente simples.
Pegue o modelo usual de regressão, , onde é o coeficiente de interesse, é um choque aleatório, as observações são indexadas por e os coeficientes são indexados por . O objetivo do MQO é minimizar a soma dos quadrados dos resíduos, ou seja . O LASSO começa dai, mas adiciona uma pequena alteração: vamos limitar o valor da soma dos valores absolutos dos coeficientes para que ele seja menor que uma constante . Ou seja, o LASSO resolve o problema:
Veja que não temos como tirar a derivada da função módulo, e portanto não podemos resolver o problema “no braço”.
Podemos reescrever o problema como um la grangeano: , onde o é o multiplicador de lagrange. Existe uma função que leva de para , então podemos ignorar o valor de e pensar só em termos de . Muitas implementações fazem isso e eu seguirei este caminho.
Veja que estamos trabalhando a soma do valor absoluto dos coeficientes. Chamamos isso de norma . Aqueles que já fizeram um curso de algebra linear conhecem a norma , conhecida como norma euclidiana: . Existe um método de estimação que usa a norma ao invés da , conhecido como ridge. Usando a norma , o problema tem solução analítica. Então, por que norma , que parece ser tão ruim de trabalhar?
Esta é uma das belezas do LASSO: a norma não gera esparsidade. A norma gera. O motivo é ilustrado na figura abaixo, tirada de Statistical Learning with Sparsity, de Trevor Hastie, Robert Tibshirani and Martin Wainwright (cujo pdf, 100% legal, você encontra aqui)
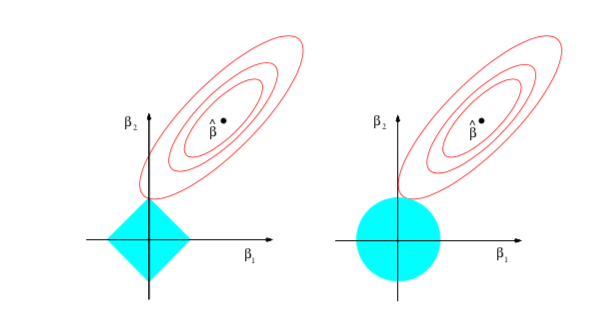
A imagem supõe que você só tem duas variáveis, por motivos ilustrativos. O é o máximo global, o conhecido estimador de MQO. Em vermelho são as curvas de nível do estimador. O que o Ridge (à direita) faz é impor uma restrição no formato de uma bola (azul). Veja que a curva de nível encosta na bola fora do eixo vertical - onde seria zero. Já o LASSO é ilustrado na figura à esquerda. Veja que, nesse caso, a curva de nível acerta na quina da restrição, efetivamente zerando o coeficiente .
Veja que, por enquanto, eu escondi o (ou o ) debaixo do tapete: eu ainda não expliquei como escolher essa variável, que de fato está no centro do problema. O pesquisador pode ter uma vaga ideia de quanto os coeficientes devem somar. Mas uma ideia exata do tamanho é complicada, então gostariamos de deixar os dados nos dizerem o certo. Existem várias estratégias, e infelizmente o glmnet só implementa uma.
Com alguma ideia do que o LASSO está fazendo, podemos colocar a mão na massa e discutir o pacote padrão para o LASSO no R, o glmnet.
##O pacote glmnet
Obviamente, começamos carregando o pacote:
## Carregando pacotes exigidos: Matrix## Loaded glmnet 4.0-2O comando para rodar uma regressão usando o LASSO é o glmnet. Ele recebe basicamente duas coisas, uma matriz com regressores e o vetor que é a variável dependente. Até ai, muito parecido com o MQO. Mas a grande diferença em termos de output é que o glmnet devolve uma matriz de coeficientes. O que o glmnet faz é estimar o modelo para um vetor de , de tal forma que você vá de nenhuma variável incluida no modelo até todas as variáveis. Isso não significa que cada estimado adicione uma variável nova ao modelo: em alguns casos o LASSO não seleciona mais ninguém, mas como ele tem mais “orçamento” para alocar, ele aumenta o valor dos coeficientes das variáveis incluídas. O glmnet resolve o problema de escolher qual usar simplesmente estimando com vários valores diferentes.
Vamos testar o glmnet gerando uma matriz com 50 variáveis e 2000 observações e criando uma variável dependente que depende só das 10 primeiras variáveis:
## 51 x 5 sparse Matrix of class "dgCMatrix"
## s0 s1 s2 s3 s4
## (Intercept) 0.0899452 0.088733817 0.08744122 0.07980104 0.07205909
## V1 . . 0.07186523 0.15461998 0.22925068
## V2 . 0.006269833 0.09268133 0.17163545 0.24390558
## V3 . . . 0.04081292 0.12344005
## V4 . 0.023074549 0.10526749 0.18520641 0.26065968
## V5 . . . 0.08775326 0.16789139
## V6 . . 0.05578103 0.13964770 0.21611625
## V7 . . 0.01023765 0.09799273 0.17949380
## V8 . 0.095583403 0.17473050 0.24967983 0.32036144
## V9 . . 0.04487896 0.12992889 0.20907527
## V10 . . . 0.06677614 0.14574878
## V11 . . . . .
## V12 . . . . .
## V13 . . . . .
## V14 . . . . .
## V15 . . . . .
## V16 . . . . .
## V17 . . . . .
## V18 . . . . .
## V19 . . . . .
## V20 . . . . .
## V21 . . . . .
## V22 . . . . .
## V23 . . . . .
## V24 . . . . .
## V25 . . . . .
## V26 . . . . .
## V27 . . . . .
## V28 . . . . .
## V29 . . . . .
## V30 . . . . .
## V31 . . . . .
## V32 . . . . .
## V33 . . . . .
## V34 . . . . .
## V35 . . . . .
## V36 . . . . .
## V37 . . . . .
## V38 . . . . .
## V39 . . . . .
## V40 . . . . .
## V41 . . . . .
## V42 . . . . .
## V43 . . . . .
## V44 . . . . .
## V45 . . . . .
## V46 . . . . .
## V47 . . . . .
## V48 . . . . .
## V49 . . . . .
## V50 . . . . .Obviamente, ainda temos que selecionar qual dos modelos nós queremos. O pacote nos permite selecionar por Cross Validation (CV). Em linhas gerais, o que o Cross Validation faz é separar os dados em blocos - 5,10, ou até mesmo blocos de uma única observação (conhecido como leave one out). Depois, escolha blocos e estime o modelo usando o glmnet. Para avaliar qual devemos usar, use o bloco que não foi usado para estimar para ver qual modelo gera o menor erro segundo alguma métrica (erro quadrático médio, por exemplo). O CV vai permitir com que cada bloco tenha uma chance de ser “fora da amostra”.
Isso pode parecer muito computacionalmente intensivo - e de fato é - mas a implementação do glmnet é tão eficiente que normalmente num piscar de olhos o modelo é estimado - no meu Dell Vostro de 2012 com i5, 6GB e Windows 10, o tempo é de apenas 0.19s. O comando que faz a seleção do é o cv.glmnet. Vamos testar no exemplo acima:
## 51 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) -0.001739634
## V1 0.937859461
## V2 0.930282416
## V3 0.908357779
## V4 0.977442350
## V5 0.929193703
## V6 0.942473364
## V7 0.953166419
## V8 0.991665138
## V9 0.960682012
## V10 0.896159947
## V11 .
## V12 .
## V13 .
## V14 .
## V15 .
## V16 .
## V17 .
## V18 .
## V19 .
## V20 -0.010307812
## V21 .
## V22 .
## V23 .
## V24 .
## V25 .
## V26 .
## V27 .
## V28 .
## V29 .
## V30 .
## V31 .
## V32 .
## V33 .
## V34 .
## V35 .
## V36 .
## V37 .
## V38 .
## V39 .
## V40 .
## V41 .
## V42 .
## V43 .
## V44 .
## V45 .
## V46 .
## V47 .
## V48 .
## V49 .
## V50 .O LASSO com CV põe apenas um coeficiente a mais. O cv.glmnet também fornece um plot com o número de coeficientes e o erro médio do Cross Validation, o que ajuda a ilustrar o ponto:
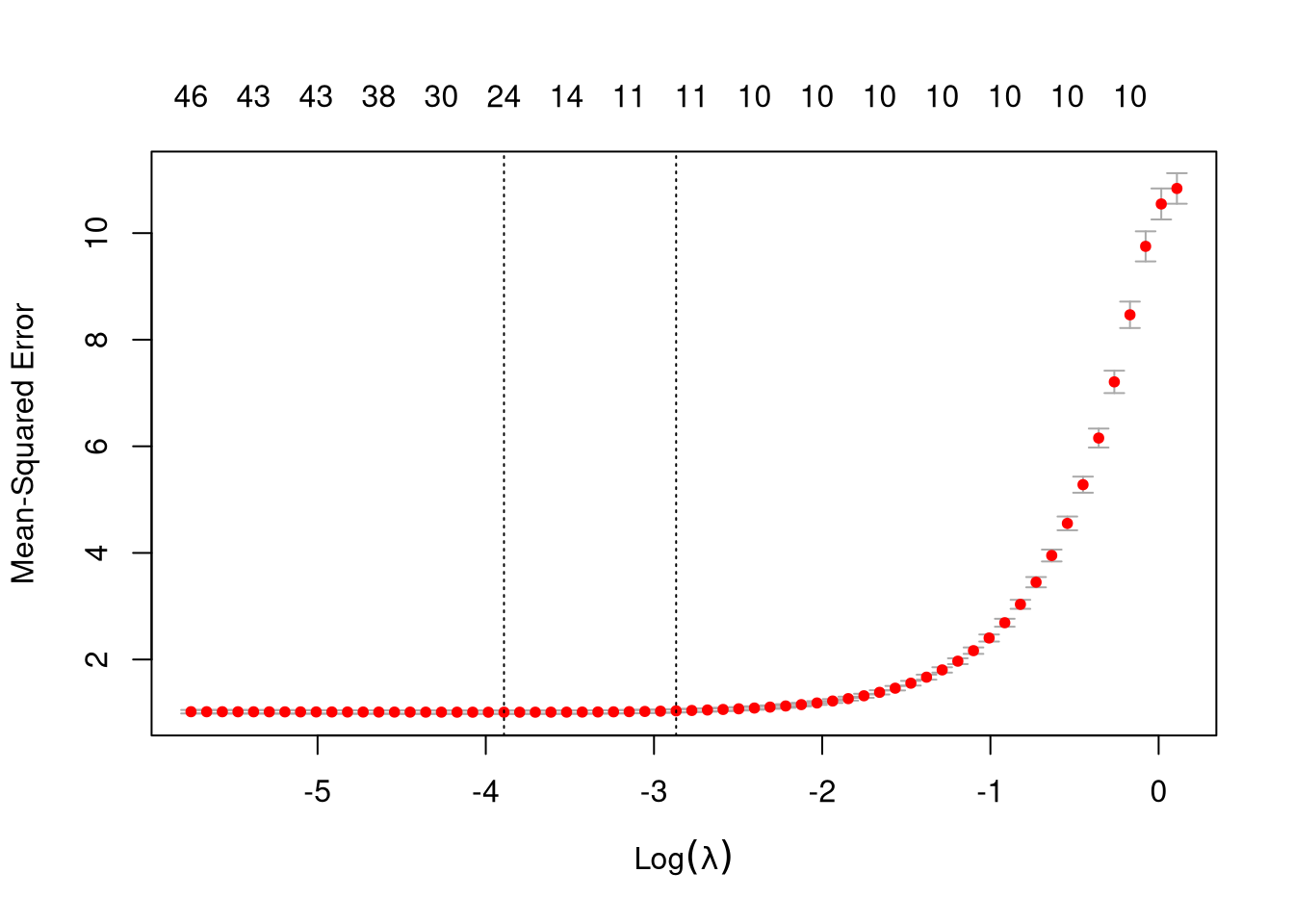
Uma das linhas tracejadas marca o número de coeficientes escolhidos: 11, número em cima. Depois dessa linha tracejada, o Mean Square Error não flutua muito. E também não há muita variação para imediatamente a direita, onde temos o modelo verdadeiro com 10 coeficientes.
(O autor agradece aos Professores Marcelo Medeiros e Pedro Souza pelas longas explicações sobre o LASSO e métodos correlatos. Pedro Cava, o outro autor deste blog, foi primordial em incentivar esse texto. Todos os erros neste texto são, como de praxe, de minha exclusiva culpa)