Amostrando de distribuições difíceis: o Markov Chain Monte Carlo
Eu recentemente tive a chance de brincar com o Markov Chain Monte Carlo (MCMC daqui por diante) no contexto de DSGE - e quando eu digo brincar eu não quero dizer que usei o Dynare, por sinal. O algoritmo é bastante esperto e funciona surpreendentemente bem. Eu não vou me atrever a entrar nos detalhes de porque funciona, mas eu vou descrever o algoritmo com algum detalhe e mostrar um exemplozinhho de regressão Bayesiana.
O algoritmo tem várias etapas e vai requerer que a gente tenha (a) uma distribuição alvo e (b) um kernel. A ideia é que é difícil retirar números aleatórios da distribuição alvo e nós queremos obter esses números por algum motivo. No caso bayesiano, eles querem obter a distribuição do parâmetro dada a regra de Bayes. Vamos temporariamente fingir que somos bayesianos. Seja os dados e os parâmetros, portanto queremos a posterior:
Onde é a prior do parâmetro - a distribuição que específica a crença do pesquisador antes de ver os dados - é a verossimelhança e é a distribuição de x. Veja que os dois primeiros elementos são fáceis de especificar; requer a distribuição de x marginalizando para o parâmetro, o que pode ser impossível de obter - se temos dez parâmetros isso requer dez integrais e nós passamos a sofrer da maldição da dimensionalidade.
Existem vários algoritmos que sorteiam de distribuições potencialmente difíceis de serem amostradas, e eu vou discutir um deles neste post. Mas todos basicamente partem da mesma ideia: sorteie números aleatórios de alguma distribuição que a gente sabe sortear. Use a densidade da distribuição que queremos de fato obter uma amostra para decidir se o parâmetro sorteado deve ser aceito ou não. Se quisermos uma estatística específica da distribuição - média, moda, mediana, desvio padrão - basta calcular a estatística empírica a partir da amostra e deixar a Lei dos Grandes Números agir.
A primeira sacada esperta é notar que, dado duas propostas de parâmetros, e , , podemos cancelar o fora. Assim, se conseguirmos uma maneira de amostrar a distribuição usando a razão, nos livramos da integral.
A segunda sacada é notar que, já que estamos usando uma razão, podemos usar o fato que se migrarmos de um ponto de menor probabilidade na posterior para um de maior probabilidade, então a razão será maior que 1.
A terceira sacada é que se a gente rejeitar toda vez que a posterior diminuir, nós teremos dois problemas:
Naturalmente o algoritmo vai convergir para o máximo e ficar preso lá
Pior, ele pode convergir para um máximo local e ficar preso lá.
Para entender o segundo ponto, dê uma olhada na seguinte função:
xx <- seq(-2,2,by = 0.05)
yy <- dnorm(xx,mean = -1,sd = 0.7) + dnorm(xx,mean=1, sd = 0.5)
plot(xx,yy, type = "l",
xlab = " ",
ylab = " ")
dm1 <- dnorm(-1, mean = -1, sd = 0.7)
d1 <- dnorm(1, mean = 1, sd = 0.5)
d0 <- dnorm(0,mean = 1, sd = 0.5) + dnorm(0, mean = -1, sd = 0.7)
points(-1, dm1, pch = 17, col = 2, cex = 2)
points(1, d1, pch = 17,col = 4, cex = 2)
points(0, d0, pch = 17,col = 3, cex = 2)
arrows(x0 = -1, y0 = dm1, x1 = 0, y1 = d0, lty = 2, lwd = 2)
arrows(x0 = 0, y0 = d0, x1 = 1, y1 = d1, lty = 2, lwd = 2)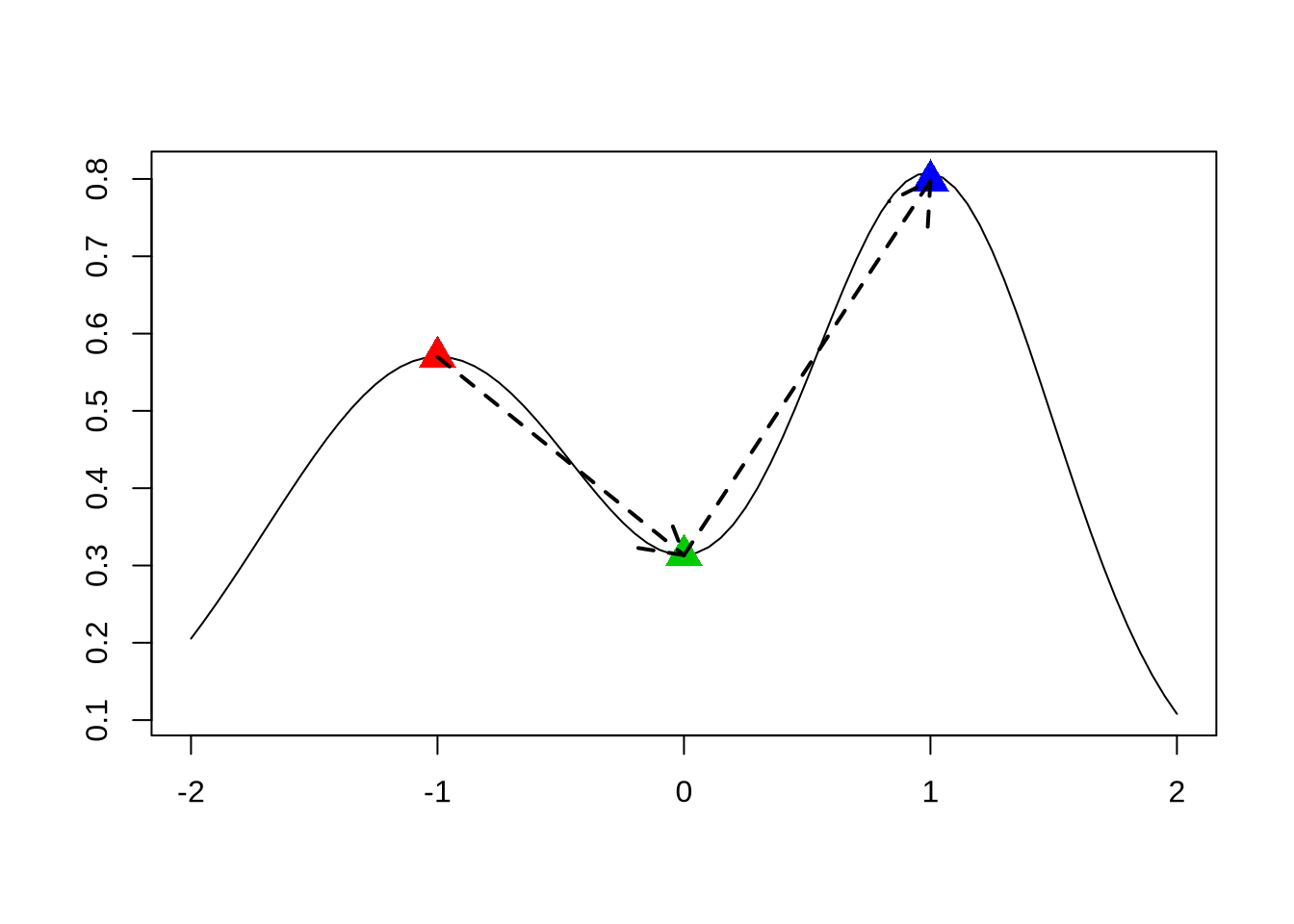
Se começamos no triângulo vermelho, qualquer ponto ao redor reduz a densidade. Mas veja que o triângulo vermelho é um máximo local e o triângulo azul o máximo global. Nós potencialmente gostaríamos de aceitar até mesmo o ponto verde se nós fizermos o caminho indicado pelas setas. Obviamente, dificilmente teremos uma situação tão evidente como essa, mas é bastante ilustrativo.
Então nós não queremos rejeitar um ponto potencial só porque ele reduz a posterior - queremos aceitar ele, de vez em quando. A ideia aqui é aceitar ele com maior probabilidade quanto mais próxima de 1 for a razão .
Espero ter convencido o leitor que é útil trabalhar com a razão para aceitar ou rejeitar um valor como representativo da distribuição. Mas ainda não atacamos o problema fundamental de como escolher esse valor. O procedimento é bem simples: escolha a distribuição kernel e sorteie um valor dela. Esse vai ser o valor que a ser testado.
A distribuição kernel tem que ser espertamente escolhida, porque existem várias condições sobre o kernel para garantir que a distribuição do algoritmo é a distribuição alvo. Para todos os efeitos, a normal centrada no último valor aceito é perfeito para os própositos - a escolha da variância é muito importante e eu vou discutir abaixo. Usar a distribuição centrada no último valor aceito gera o algoritmo chamado Random Walk Metropolis Hasting - a parte do Random Walk se deve ao fato da distribuição do sorteio ser centrado no último valor aceito.
Nós também vamos querer corrigir a probabilidade de aceitação e rejeição pelo kernel. A intuição é que não só importa o quão provável é o novo parâmetro dado a posterior, mas o quão provável é o parâmetro dado o kernel: se nós sorteamos um parâmetro na ponta do kernel isso tem que ser levado em conta. Uma explicação um pouco mais séria: o importante é que a probabilidade de passar do ponto x para o ponto y seja igual. Uma distribuição qualquer não garante isso, mas corrigir a razão pelo kernel faz com que isso seja satisfeito. Isso não significa que qualquer distribuição serve como Kernel. Então, seja a probabilidade de passar do ponto para . A razão que vamos nos preocupar é:
Veja que se o kernel faz com que a probabilidade de passar do ponto x para o ponto y é igual a de passar do ponto y para o ponto x, então = , os termos se cancelam e voltamos a nossa razão que eu coloquei no começo.
Eu escrevi bastante coisa e parece justo fazer um resumo do passo a passo do discutido até aqui:
Defina a função kernel, a posterior (que depende da verossimelhança e das priors) e um ponto inicial do espaço paramétrico,
- Sorteie um possível valor possível do parâmetro da distribuição kernel. Chame esse valor de
- Compute
- Sorteie u ~ Uniforme(0,1)
- Se , aceite e . Caso contrário, rejeite e
- Itere n vezes
Veja que se o parâmetro for rejeitado, o valor antigo vai ser repetido na simulação. Isso vai garantir com que pontos de alta probabilidade apareçam mais na distribuição empírica, e portanto tenha probabilidade empírica maior.
Um exemplo
Como de praxe, um exemplo vai ajudar muito a entender tudo que eu escrevi. Vamos fazer uma situação super simples onde eu tenho uma regressão e , 100 observações e
x <- rnorm(100)
y <- 1.5*x + rnorm(100)Vamos fazer a função de verossimelhança. Como o erro é normal, então . Eu vou escrever direto a log verossimelhança
loglike <- function(y,x,beta){
sum(dnorm(y-beta*x,log = T))
}Para a prior, eu vou colocar uma Normal de média 0 e desvio padrão 3, e com isso temos a posterior:
posterior <- function(y,x,beta){
loglike(y,x,beta) + dnorm(beta,mean=0,sd=3,log = T)
}Note que como eu estou trabalhando com o log de tudo, ao invés de multiplicar a verossimelhança pela prior, eu somo as duas. Há bons motivos para trabalhar com a soma ao invés de multiplicação: thou shall not multiply two small numbers é um dos mandamentos de computação numérica (juro que escrevo um post curtinho sobre ponto flutuante!).
Agora sim podemos passar para o MCMC. Eu vou fazer 5000 passos e iniciar em zero:
set.seed(19965)
n <- 10000
params_mcmc <- rep(0,n)
for(i in 2:n){
par_novo <- rnorm(1,mean = params_mcmc[i-1], sd = 0.5)
kernel_velho <- dnorm(par_novo,mean = params_mcmc[i-1], sd = 0.5, log = T)
kernel_novo <- dnorm(params_mcmc[i-1],mean = par_novo, sd = 0.5, log = T)
r_denom <- posterior(y,x,par_novo) + kernel_novo
r_nomi <- posterior(y,x,params_mcmc[i-1]) + kernel_velho
r <- r_denom - r_nomi
u <- runif(1)
r <- max(exp(r),0)
if(r > u){
params_mcmc[i] <- par_novo
} else{
params_mcmc[i] <- params_mcmc[i-1]
}
}Usualmente, demora algum tempo até a cadeia de markov convergir para a distribuição que queremos - isso depende uma série de coisas, como o quão perto é o nosso chute inicial da moda e a complexidade do modelo. Em geral, se descarta algumas observações iniciais enquanto a cadeia ainda está convergindo, que é chamado burn-in. Eu vou jogar as 4 mil primeiras fora e ver o histograma da distribuição:
library(ggplot2)
pars <- params_mcmc[4001:10000]
ggplot(data.frame(pars = pars),aes(pars)) + geom_histogram(bins = 30) + scale_x_continuous(breaks = seq(1,2,by = 0.1)) + theme_bw() 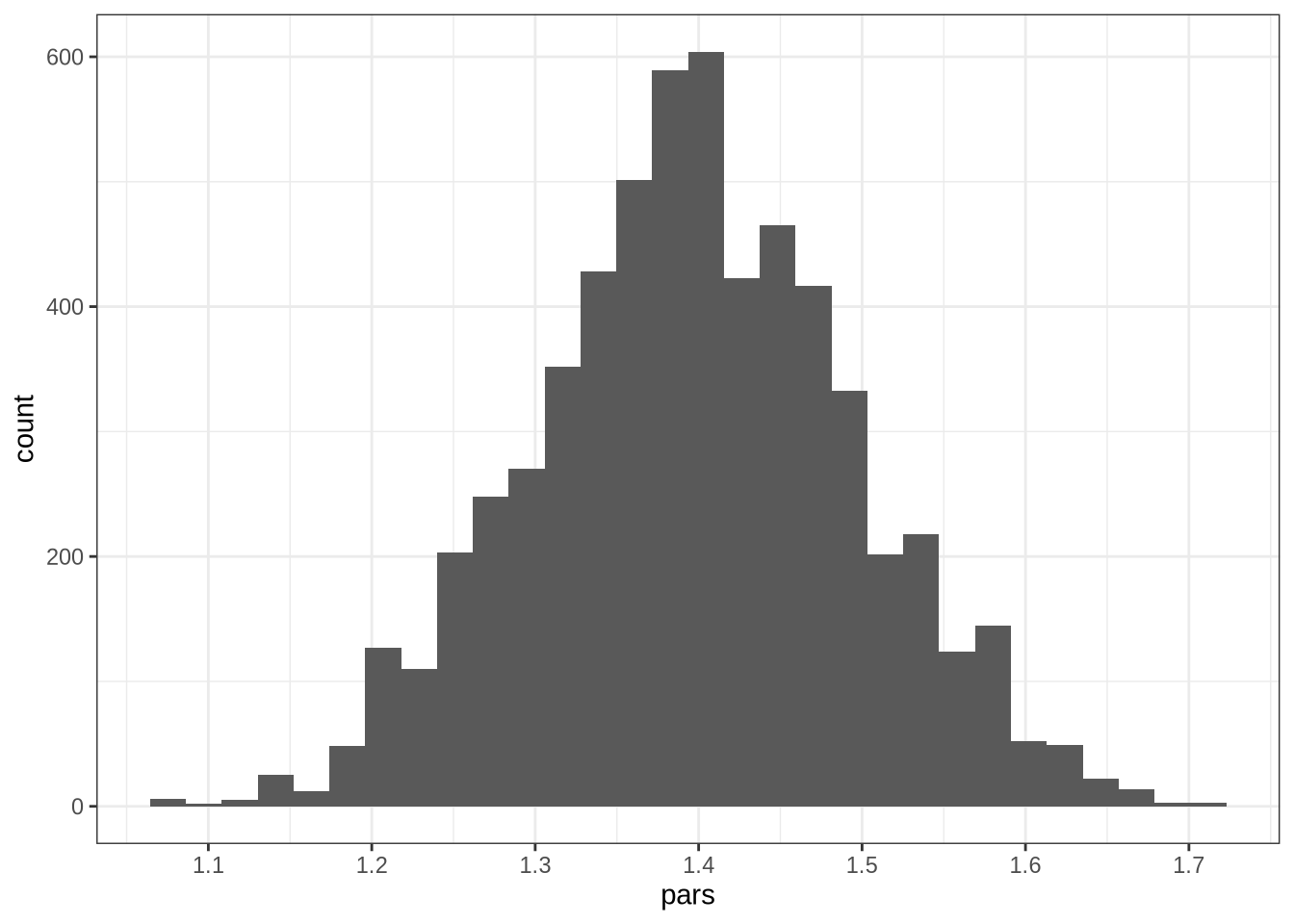
Veja que o histograma não é muito suave e nem está centrado no valor verdadeiro do parâmetro. Temos vários possíveis suspeitos, mas eu imagino que o problema seja que nós temos apenas 100 observações. Observe que se rodarmos o bom e velho Mínimos Quadrados, obtemos o coeficiente 1.3973191 - próximo da moda do histograma. Vamos aumentar o tamanho da amostra e repetir o MCMC:
x <- rnorm(1000)
y <- 1.5*x + rnorm(1000)
n <- 10000
params_mcmc <- rep(0,n)
aceitacao <- 0
for(i in 2:n){
par_novo <- rnorm(1,mean = params_mcmc[i-1], sd = 0.5)
kernel_velho <- dnorm(par_novo,mean = params_mcmc[i-1], sd = 0.5, log = T)
kernel_novo <- dnorm(params_mcmc[i-1],mean = par_novo, sd = 0.5, log = T)
r_denom <- posterior(y,x,par_novo) + kernel_novo
r_nomi <- posterior(y,x,params_mcmc[i-1]) + kernel_velho
r <- r_denom - r_nomi
u <- runif(1)
r <- max(exp(r),0)
if(r > u){
params_mcmc[i] <- par_novo
aceitacao <- aceitacao + 1 #continue lendo, isso vai ficar claro
} else{
params_mcmc[i] <- params_mcmc[i-1]
}
}
pars <- params_mcmc[4000:10000]
ggplot(data.frame(pars = pars),aes(pars)) + geom_histogram(bins = 30) + scale_x_continuous(breaks = seq(1,2,by = 0.05)) + theme_bw() 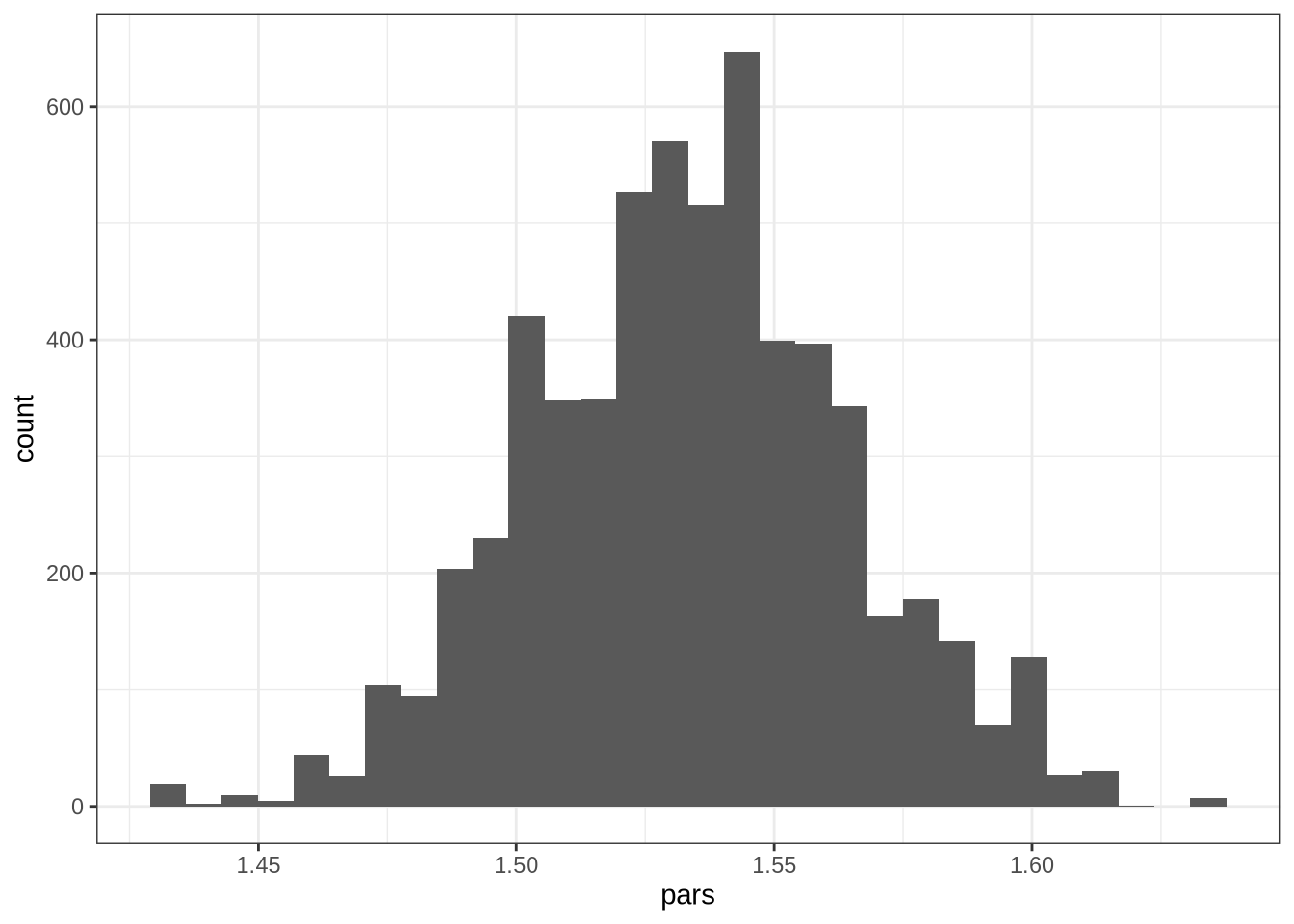
Agora o histograma nos mostra bem perto de 1.5!
Veja que eu falei que a variância do Kernel é bem importante. Em geral, a variância é escolhida de maneira que a aceitação fique perto dos 20% - esse é o valor ótimo de aceitação para o MCMC. Eu ignorei totalmente esse fato por enquanto, mas observe que eu salvei uma variável que nos dá a taxa de aceitação: é a variável aceitacao(duh); basta dividir ela pelo total de simulações (n) e teremos nossa taxa de aceitação. No caso, ela é 8.16%. Vamos aumentar a variância e ver o que acontece:
x <- rnorm(1000)
y <- 1.5*x + rnorm(1000)
n <- 10000
params_mcmc <- rep(0,n)
aceitacao <- 0
for(i in 2:n){
par_novo <- rnorm(1,mean = params_mcmc[i-1], sd = 1.5)
kernel_velho <- dnorm(par_novo,mean = params_mcmc[i-1], sd = 1.5, log = T)
kernel_novo <- dnorm(params_mcmc[i-1],mean = par_novo, sd = 1.5, log = T)
r_denom <- posterior(y,x,par_novo) + kernel_novo
r_nomi <- posterior(y,x,params_mcmc[i-1]) + kernel_velho
r <- r_denom - r_nomi
u <- runif(1)
r <- max(exp(r),0)
if(r > u){
params_mcmc[i] <- par_novo
aceitacao <- aceitacao + 1
} else{
params_mcmc[i] <- params_mcmc[i-1]
}
}
pars <- params_mcmc[2000:10000]
ggplot(data.frame(pars = pars),aes(pars)) + geom_histogram(bins = 30) + scale_x_continuous(breaks = seq(1,2,by = 0.05)) + theme_bw() 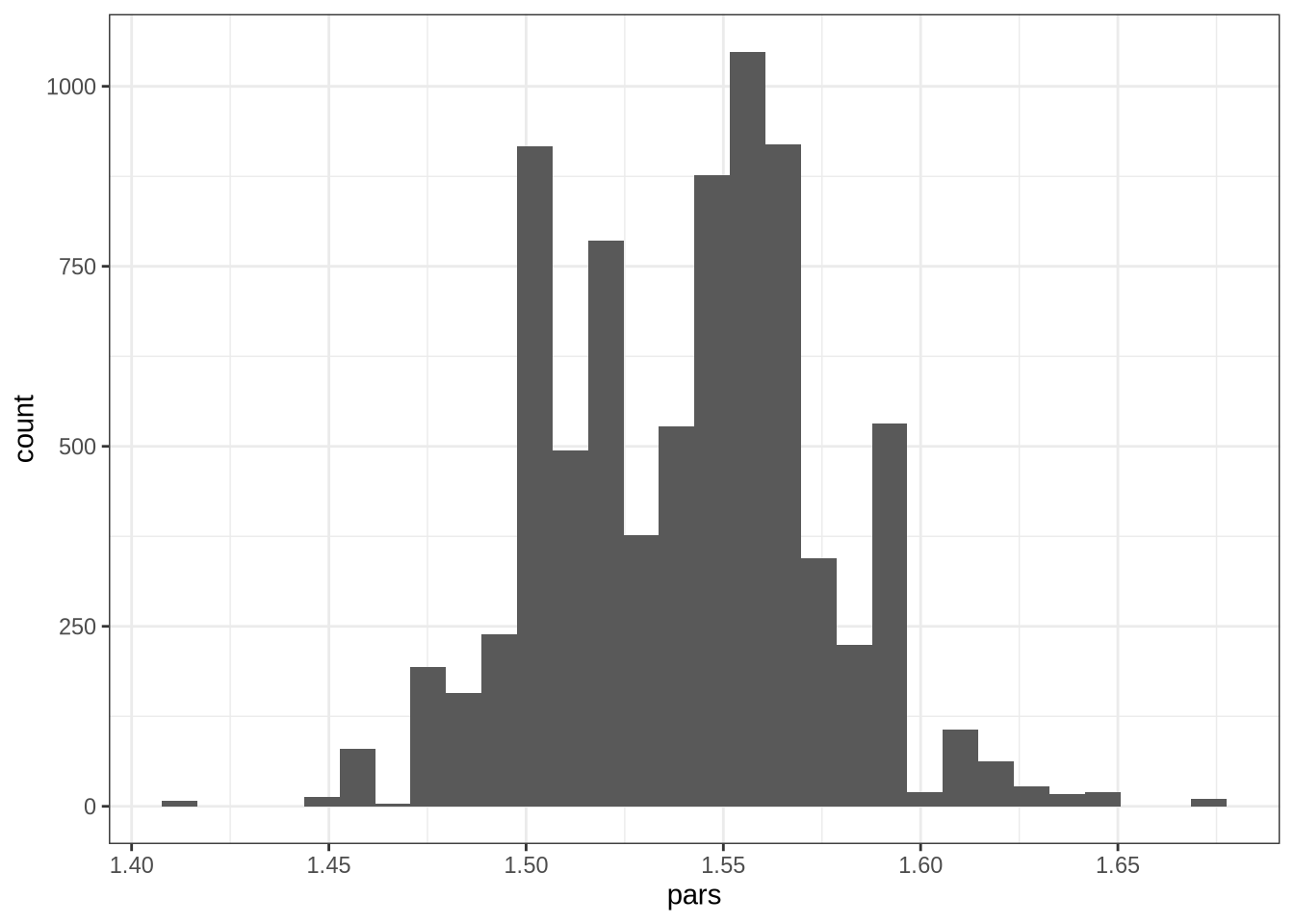
A taxa de aceitação agora é 3.39%. variância muito alta faz a distribuição ficar esquisita. A intuição é simples: como é um passeio aleatório, a distribuição vai passear por uma região muito maior, possivelmente visitando pontos com uma probabilidade muito baixa de ser aceita.
Podemos ter o problema oposto: a variância ser muito baixa e o algoritmo demorar um tempo enorme para visitar a distribuição:
x <- rnorm(1000)
y <- 1.5*x + rnorm(1000)
n <- 10000
params_mcmc <- rep(0,n)
aceitacao <- 0
for(i in 2:n){
par_novo <- rnorm(1,mean = params_mcmc[i-1], sd = 1e-8)
kernel_velho <- dnorm(par_novo,mean = params_mcmc[i-1], sd = 1e-8, log = T)
kernel_novo <- dnorm(params_mcmc[i-1],mean = par_novo, sd = 1e-8, log = T)
r_denom <- posterior(y,x,par_novo) + kernel_novo
r_nomi <- posterior(y,x,params_mcmc[i-1]) + kernel_velho
r <- r_denom - r_nomi
u <- runif(1)
r <- max(exp(r),0)
if(r > u){
params_mcmc[i] <- par_novo
aceitacao <- aceitacao + 1
} else{
params_mcmc[i] <- params_mcmc[i-1]
}
}
pars <- params_mcmc[2000:10000]
ggplot(data.frame(pars = pars),aes(pars)) + geom_histogram(bins = 30) + theme_bw() 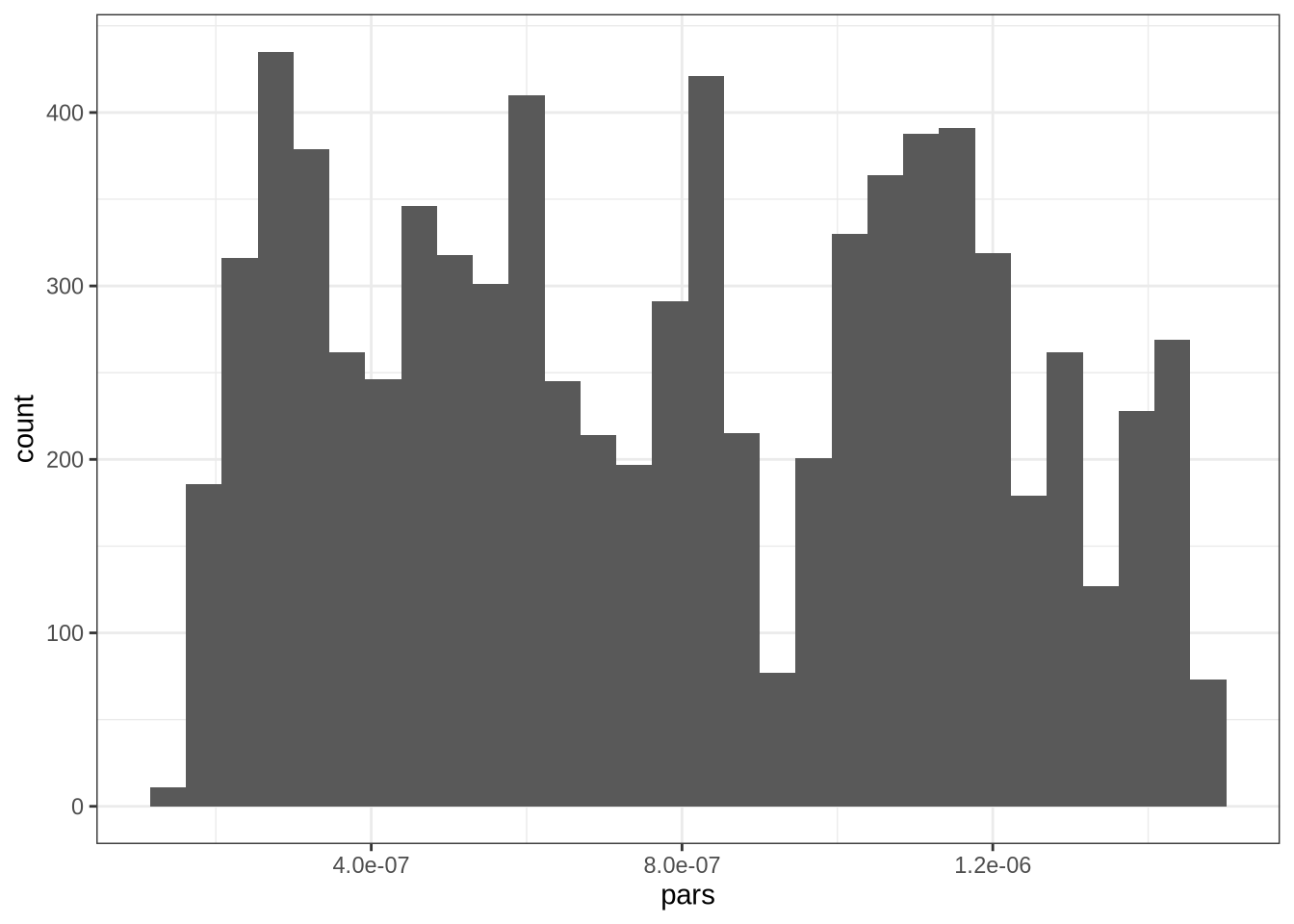
Na prática, como colocamos 10 mil passos, a distribuição fica uma bagunça perto de zero. Ou seja, errar a variância tem efeitos perturbadores no algoritmo. A taxa de aceitação é 99.99%.
Veja que isso sugere que se nós estivermos aceitando demais, nós devemos aumentar a variância do kernel; e se tivermos aceitando de menos devemos diminuir a variância.
Isso ilustra como MCMC funciona. Longe de ser uma caixa preta, é um algoritmo relativamente simples, fácil de implementar. A taxa de aceitação ótima e o tamanho do burn-in são problemáticos, com modelos grandes exigindo um milhão de passos e quase a metade sendo descartados (!). Ainda temos um parâmetro para escolher, a variância do Kernel. Veja que ainda temos uma limitação que eu nem toquei: como a distribuição é Normal, os parâmetros devem ser definidos nos Reais. É comum em modelos, inclusive modelos DSGE, definir um pedaço dos reais para os parâmetros - a taxa de desconto intertemporal está entra 0 e 1, por exemplo. Nesse caso, temos que fazer transformações nos parâmetros e alterar a distribuição via o jacobiano… um pouco mais confuso! Mas os fundamentos não mudam.
A ideia de passear aleatoriamente pelo espaço paramétrico não é tão brilhante quando temos uma dimensão muito alta: é como tentar procurar a chave perdida andando por ai e torcendo para topar com ela em algum momento. Os usuários de métodos de MCMC se deram conta disso e desenvolveram métodos que ao invés de usar um passeio aleatório passeiam pelo espaço paramétrico de maneira um pouco mais esperta…
Um excelente livro sobre MCMC e algoritmos correlatos é o Bayesian Data Analysis, de Andrew Gelman e (muitos) coautores.