{tidyverse}, Simulações e Processamento de Séries Temporais
Só para deixar tudo bem claro quanto ao que eu quero dizer quando falo em uma função impura: se fornecida os mesmos argumentos devolverá o mesmo resultado. É simples construir alguns exemplos.
foo1 <- function(x) {
lubridate::now() + lubridate::dseconds(x + sample(-10:10, size = 1))
}
foo1(1)## [1] "2020-12-11 07:03:25 -03"foo1(1)## [1] "2020-12-11 07:03:22 -03"foo1(1)## [1] "2020-12-11 07:03:40 -03"Funções impuras também podem assim ser porque desencadeiam efeitos colaterais, como por exemplo escrever algum arquivo na memória.
foo2 <- function(x) {
file.create(glue::glue('{x}.txt'))
}Isso não é necessariamente ruim, o objetivo final de código em produção é desencadear efeitos colaterais, mas isso leva à código com consequências menos claras e por isso eu aprendi nos manuais e com os erros a evitar impureza. Pelo exercício vou tentar ilustrar uma aplicação onde impureza traz ergonomia.
É bem comum que eu simule dados de processos estocásticos aqui. Um AR1 com \(\beta = 1\), por exemplo:
library(tidyverse)
N <- 1000
(data <- tibble(t = 1:N,
y = accumulate(rnorm(N), ~ .x + .y)))## # A tibble: 1,000 x 2
## t y
## <int> <dbl>
## 1 1 0.229
## 2 2 1.68
## 3 3 1.94
## 4 4 3.28
## 5 5 3.28
## 6 6 3.03
## 7 7 4.01
## 8 8 4.09
## 9 9 3.75
## 10 10 6.03
## # … with 990 more rowsdata %>%
ggplot(aes(x = t, y = y)) +
geom_line(size = 1.2, color = 'red') +
theme_minimal() +
labs(x = 't',
y = '')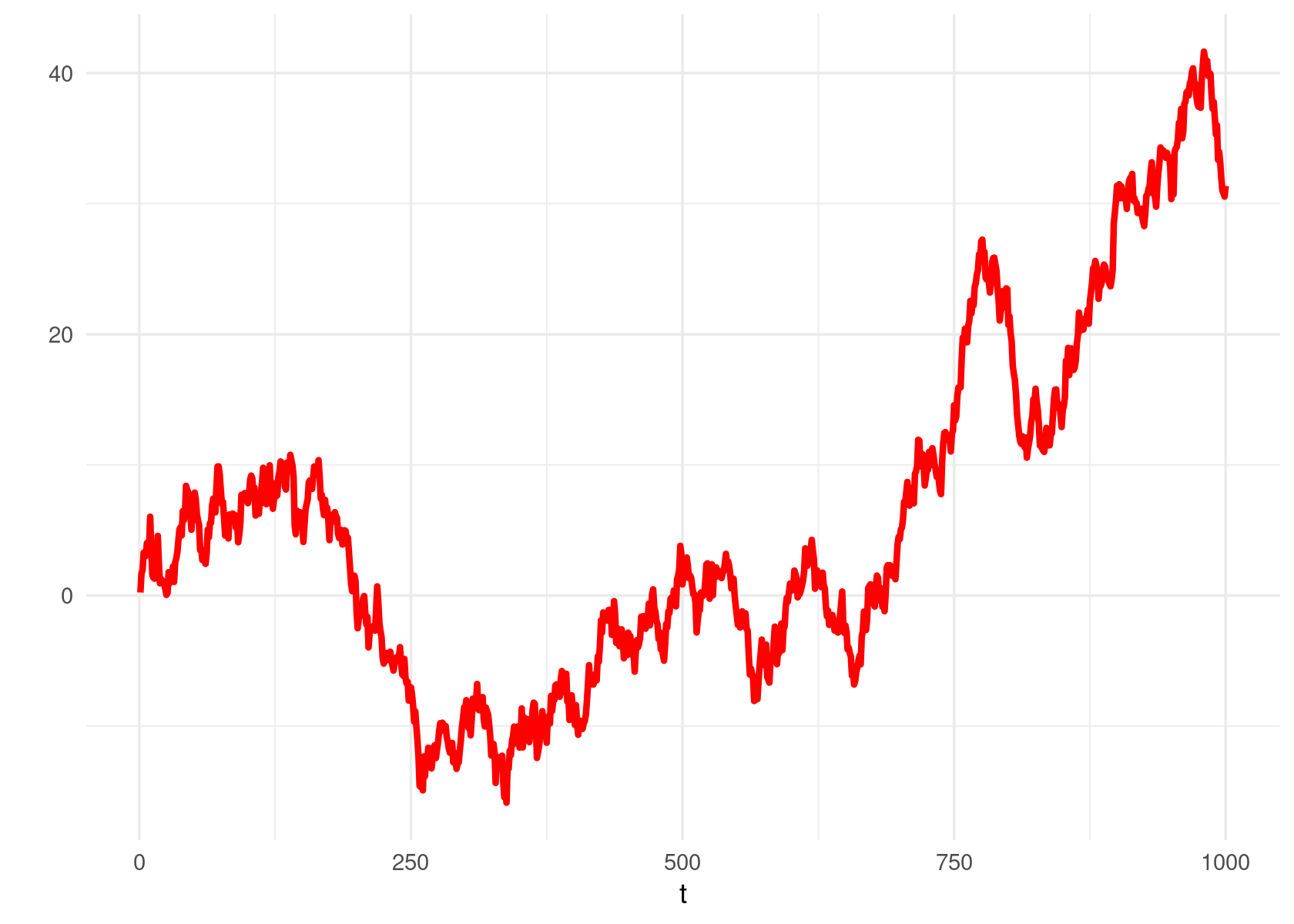
E tem duas coisas aí que eu gostaria de mudar:
- Referência explícita e repetida ao tamanho da amostra, que é sempre igual e dado pelo tamanho do tibble, contextual
- Me referir ao processo gerador com alguma abstração, ao invés de explicitamente simular choques e soma-los.
Podemos resolver isso tudo em uma função que (i) sabe que precisa gerar um AR1 com certo \(n\) e (ii) é agnóstica em relação à distribuição dos choques.
Primeiro, descobrir se a função foi chamada em um contexto de dados, dentro de um tibble. A ideia aqui é olhar como se comporta dplyr::n, que traz a informação contextual do tamanho do tibble usado como ambiente quando foi executada. Se não foi chamada dentro de um tibble, vai retornar erro:
n()## Error: `n()` must only be used inside dplyr verbs.tibble(A = rnorm(100, mean = -1),
sinal = A > 0) %>%
group_by(sinal) %>%
summarise(N = n())## # A tibble: 2 x 2
## sinal N
## <lgl> <int>
## 1 FALSE 81
## 2 TRUE 19Dito isso, precisamos capturar o erro, caso ocorra, então é bom embrulhar a função em algum advérbio do purrr.
data_context <- function() {
foo <- purrr::possibly(dplyr::n, otherwise = NULL)
if(rlang::is_null(foo())) {
return(FALSE)
} else {
return(TRUE)
}
}E agora a função que simula o processo em si.
ar <- function(
.distfn = rnorm,
...,
.k = 1L,
beta = runif(n = .k),
.init = 0,
.n = NULL) {
# Guardião -------------------------
if(length(beta) != .k) {
rlang::abort('Parameter vector must have one ')
}
if(rlang::is_null(.n) & !data_context()) {
rlang::abort('Function called outside a tibble, provide .n argument.')
}
if(data_context()) {
n <- dplyr::n()
} else {
n <- .n
}
# Computando as inovações ------------------
params <- list(..., n = n)
innovations <- rlang::exec(.distfn, !!!params)
# gerando os lags e computando os valores ----------------
as.list(1:.k) %>%
purrr::set_names(1:.k) %>%
purrr::map_dfc(~ dplyr::lag(innovations, .x)) %>%
dplyr::rowwise() %>%
dplyr::mutate(
total =
sum(
beta*dplyr::c_across(tidyselect::everything()),
na.rm = TRUE) +
.init) %>%
dplyr::pull(total)
}E agora podemos fazer coisas como isso:
plot(ar(.n = 100))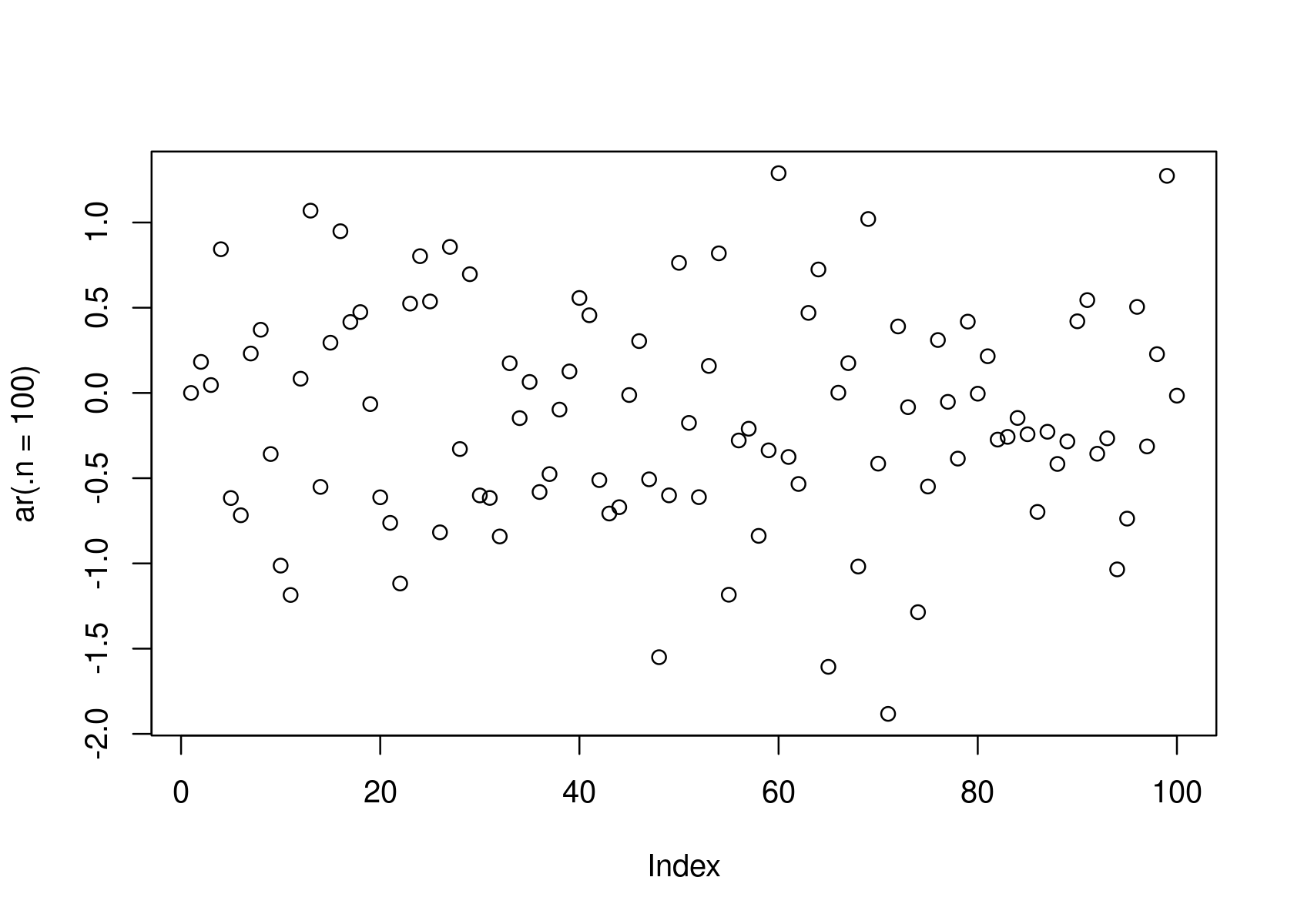
tibble(t = 1:100) %>%
mutate(
y1 = ar(sd = .2, beta = .7),
y2 = ar(runif, min = -1, .k = 10, beta = runif(10, max = .1))) %>%
pivot_longer(-t, names_to = 'processo') %>%
ggplot(aes(x = t, y = value, color = processo, group = processo)) +
geom_line(size = 1.2) +
theme_minimal()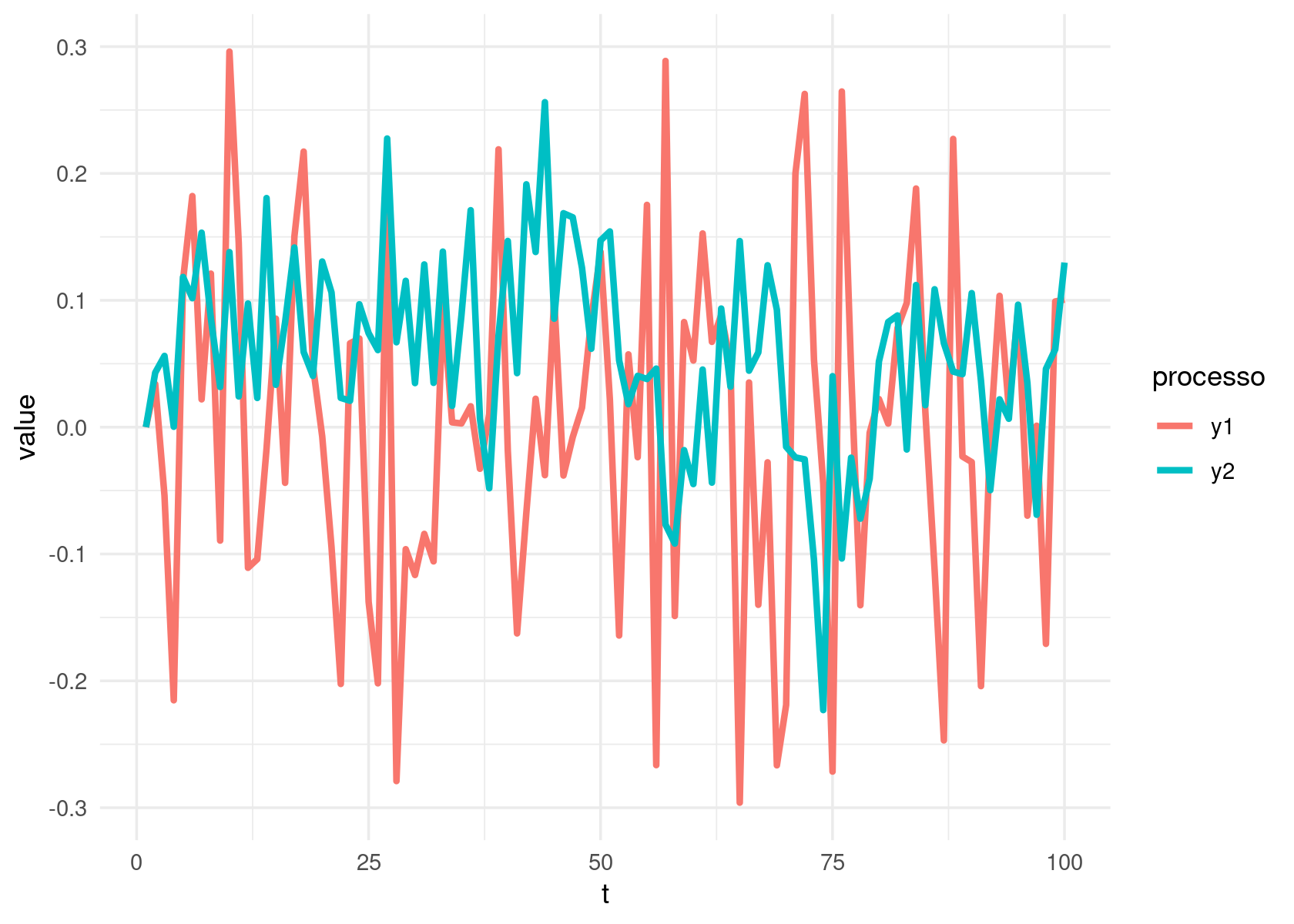
Um próximo passo seria extrair alguns momentos ao longo do tempo nesses processos. Para isso não precisamos implementar soluções, o slider dá conta. É uma espécie de purrr para operações em janelas móveis. Nele a prima mais próxima de purrr::map é slider::slide, que também tem variantes tipadas com terminações em _lgl, _dbl e etc.
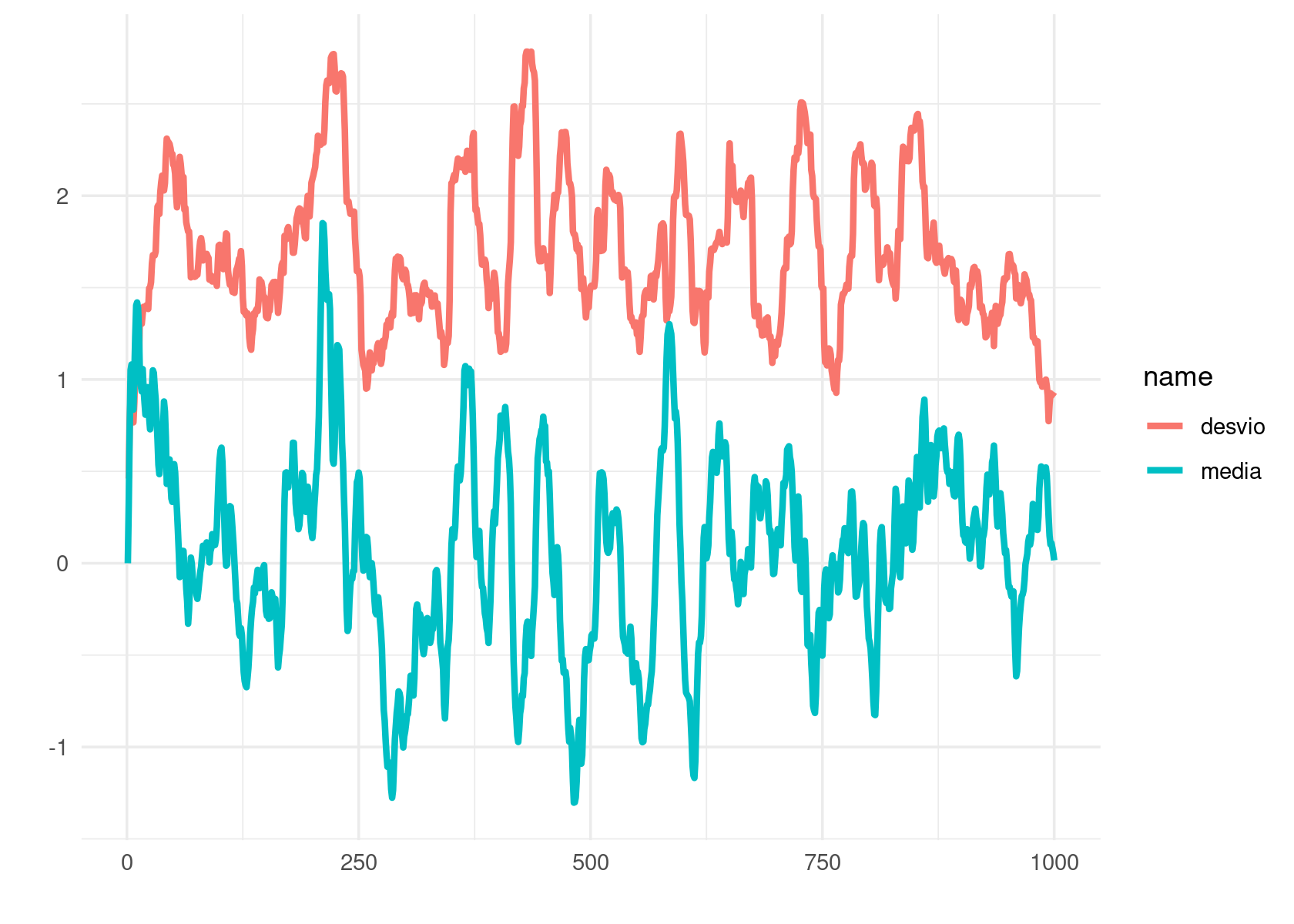
library(slider)
tibble(t = 1:1000) %>%
mutate(y = ar(.k = 3, beta = rep(1, times = 3)),
media = slide_dbl(y, mean, .before = 25),
desvio = slide_dbl(y, sd, .before = 25)) %>%
pivot_longer(media:desvio) %>%
ggplot(aes(x = t, y = value, color = name, group = name)) +
geom_line(size = 1.2, aplha = .9) +
theme_minimal() +
labs(x = '',
y = '')