R mais rápido
Os problemas de velocidade do R são muito conhecidos. Já foram feitos vários esforços para acelerar a linguagem no base-R, colocando Just In Time Compilation, por exemplo. Mesmo assim a linguagem ainda é relativamente lenta.
Existem várias iniciativas para acelerar o R. Uma das mais famosas é o R da Microsoft, o R Open. Eles usam bibliotecas que agilizam as contas e usa vários processadores sem precisar fazer nenhum setup.
Uma outra opção, absolutamente obscura, é o pqR - pretty quick R. Absolutamente obscuro e prometendo mais velocidade é uma boa receita para ter meu interesse.
O pqR é um pouco mais ousado porque envolve reescrever trechos do R e implementar novas ferramentas dentro do R. Antes de discutir o lado bom, deixa eu levantar alguns alguns obstáculos:
- Você tem que compilar
- Ele é baseado no R 2.5
O 2 significa que você pode instalar pacotes porque o pqR tem o “próprio CRAN” - mas se eles não existiam há anos atrás, não vai ter. E mesmo se tiver ele pode não ter vários comandos que você se acostumou a usar.
O 1 é em certos aspectos mais sério - e em outros menos. Compilar significa que efetivamente você vai ter que gerar o software dos arquivos que definem ele - nada de dar dois cliques e continuar. Isso também aumenta as chances de dar problema durante o processo. A vantagem é que compilar permite o programa rodar mais rápido.
Dado isso, eu vou dizer porque você talvez se interesse no pqR e depois explicar como eu compilei.
The good
O pqR foi amplamente reescrito pelo Radford Neal para agilizar o processamento - na página do pqR vocês podem ler algumas das mudanças. Como de praxe, o importante é saber se isso faz diferença, especialmente para as versões mais recentes e maneiras mais eficientes de fazer as coisas, como o purrr.
Como eu faço muita simulação, que é bastante intensivo computacionalmente, eu escolhi elas como benchmark. Tudo foi rodado no mesmo computador, um Dell Vostro com i5 e 6 GB de RAM, e usando arch linux e o RStudio. Para medir o tempo eu vou usar o tictoc. Eu dei duas chances pra todo mundo e fiquei com a melhor.
Eu vou separar em casos e apresentar os resultados em uma tabela mais embaixo:
O bom e velho testar a qualidade de seleção de modelo usando o LASSO, que segue muito de perto um post que eu fiz: é o código desse post repetido 2000 vezes. Tanto no R 4.0.2 quanto no pqR eu usei o for.
Simulação para obter a distribuição de estatísticas em casos não estacionários, que envolvem repetir a mesma coisa 100 mil vezes: estimar dois modelos lineares e uma pancada de contas de multiplciar, somar e dividir.
Nesse caso 2 eu dei uma colher de chá pro R normal - porque eu vi o resultado do caso 1 - e fiz com o purrr, que é mais rápido que o for e não está disponível pro pqR.
O terceiro exemplo é cortesia do Twitter: a galera de econometria espacial me disse que “rasterização de polígonos” é intensivo e eu achei que era uma boa testar. O primeiro pacote que me sugeriram não tinha para o pqR, mas o raster tinha. Eu rodei o seguinte código, baseado em um exemplo do pacote:
library(raster)
r <- raster(nrow=1440, ncol=1440)
r[] <- runif(ncell(r)) * 10
r[r>8] <- NA
pol <- rasterToPolygons(r, fun=function(x){x>6})O maior problema aqui foi memória RAM: esse exemplo é relativamente rápido mas ocupou uns 6.5 GB de memória! Fui informado que o pessoal usa computador com mais de 5 vezes o RAM que eu tenho - e não é o suficiente. O tempo em segundos dos três casos:
| Caso | pqR | R 4.0.2 | Razão |
|---|---|---|---|
| 1 | 276.67 | 499.35 | 1.80 |
| 2 | 875.45 | 1414.22 | 1.60 |
| 3 | 50 | 71 | 1.42 |
O caso 1 é uma baita surra e me fez passar a roubar para o R 4.0.2. Pro caso 2, não adiantou muito. O terceiro exemplo a diferença não foi tão grande assim porque eles foram rápidos, e é o caso em que o pqR deu o menor ganho. Ainda assim, se isso for a diferença entre um dia ou 16h, eu acho que pode ser um ganho bastante importante. Uma coisa importante é que nesse exemplo é possível que as mudanças no garbage collection do pqR faça diferença. Eu não testei isso porque eu não tenho tanta experiência com esse tipo de problema e, mais importante, porque isso encheu a memória ram e a solução foi reiniciar.
Levem em consideração que eu não compilei o R 4.0.2, e compilar o R deve aumentar a velocidade dele. Mas eu também não compilei o pqR da maneira mais eficiente possível, então ainda deve dá pra ganhar uns segundos no pqR. Se toda diferença se deve ao fato que eu compilei um e não o outro - o que eu não acho que seja verdade, mas eu posso estar errado - então eu passarei a compilar mais software.
(Na página do pqR tem alguns testes comparando com o R 3.0.1, que podem ser interessantes)
Outra coisa legal do pqR é que na versão mais recente - a que ainda está em teste - tem automatic differentiation (ou auto diff, para os íntimos). Auto diff é uma tentativa de ser mais preciso e mais rápido que diferenciação numérica (diferenças finitas) e mais rápido que diferenciação simbólica. O Julia tem e é muito usado lá. O tensorflow também implementa auto diff. Como eu já discuti em um post anterior, o R não tem um pacote que faça isso nativamente - o task view de matemática numérica sugere um pacote do GitHub (!) que usa o Julia para calcular as derivadas (!!) e devolve pro R.
Só para dar um exemplo, vamos estimar alguma coisa por máxima verossimilhança. Vai ser uma normal com 50 variáveis e 100 observações. Eu vou escrever a função de máximia verossimillhança e usar o optim para achar o ótimo usando BFGS, que requer o gradiente. Eu vou rodar o comando duas vezes no pqR, uma fornecendo o gradiente para o optim usando automatic differentation e a outra eu vou deixar rodar sem fornecer o gradiente, então o R automaticamente usa diferenças finitas. O código:
library(tictoc)
log_mvn <- function(x,mu,S){
m <- length(mu)
return(-m/2*log(2*pi) -1/2*log(det(S)) -1/2*t(x-mu)%*%solve(S)%*%(x-mu))
}
par <- runif(50,-1,1)
x <- matrix(rnorm(100*50),ncol = 50)
y <- x%*%par + rnorm(100)
ll <- function(par) -1*log_mvn(y,x%*%par,diag(100))
grad <- function(par) attr(with gradient(par=par) ll(par),"gradient")
tic()
otim_auto <- optim(rep(0,50),ll,grad, method = "BFGS")
toc()
tic()
otim <- optim(rep(0,50),ll, method = "BFGS")
toc()
Veja que esse código não vai rodar no R normal. O comando de gradiente é realmente esquisito e eu embalei ele numa função. As diferenças de qualidade da estimação não são visíveis:
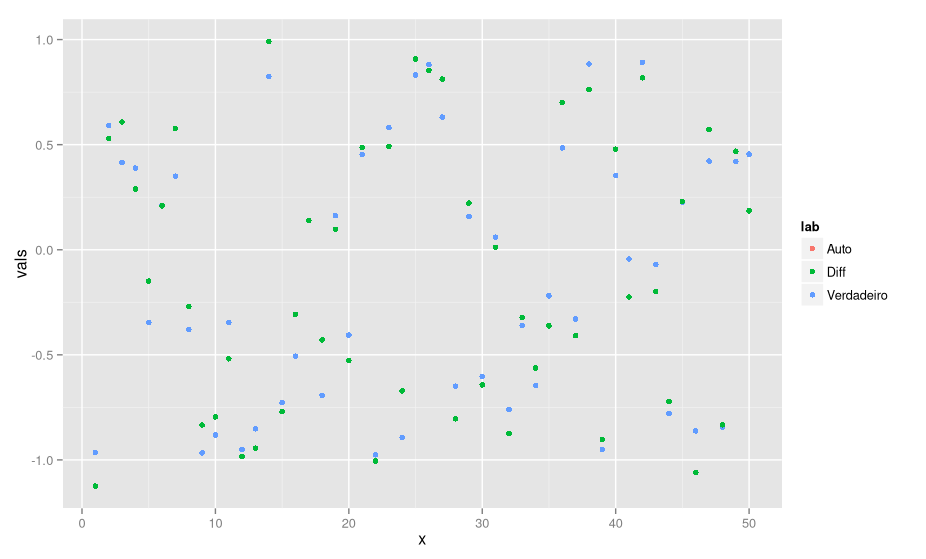
Você não vê nenhum ponto do auto diff porque o caso de diferenças finitas cobre ele. Mas a diferença de tempo é estúpida: com o auto diff isso demora 0.33s e com diferenças finitas 6.3s!
The bad
O fato de depender do R 2.5 é ruim especialmente porque qualquer projeto que use pacotes ou até mesmo comandos que foram implementados depois dessa versão estarão indisponíveis. Pacotes que dependem de programas externos vão sofrer ainda mais dependendo das mudanças no programa externo ou no pacote. A solução mais simples é manter duas cópias do R, uma do 4.0.2 e a outra do pqR. Isso vai exigir um certo malabarismo no Windows se você quiser usar o RStudio para os dois. No Linux é um pouco mais fácil, como eu explico a seguir. Mesmo assim, escrevendo esse post eu precisei fazer um certo malabarismo para rodar coisas no pqR, voltar para o Rstudio com o R 4.0.2, colocar os resultados no post e gerar o blog.
The ugly
Bom, vamos falar de compilar o pqR. Eu sou obrigado a fazer duas observações:
- Eu não fiz computação, então é uma explicação do que eu entendo - provavelmente o pessoal de computação vai ter pequenos ataques cardíacos lendo o que vem a seguir. Espero não ter nada extremamente errado.
- Isso é um pouco mais avançado do que a média, em certos aspectos. As chances de bater cabeça e perder um tempo com isso são altas. Então talvez você não queira fazer isso se você não está muito confortável em programar ou não quer bater cabeça.
Veja que compilar vai variar de sistema para sistema, e até mesmo variações dentro de famílias - i.e. distros do Linux diferentes ou parques nacionais da Apple diferentes.
Obviamente, o guia de instalação do pqR é o padrão ouro - isso aqui é nota de rodapé (mas leia o paragráfo seguinte). Eu vou reproduzir todos os passos para deixar o mais claro possível. O que eu fiz foi pro EndeavourOS, e deve funcionar bem pra quem tem arch e deve ajudar outras distros de Linux.
Uma coisa importante: se você está usando Linux, comece checando a sua versão do gcc indo no terminal e fazendo gcc –version. Se a versão for a 10, leia as instruções a seguir porque todos os problemas que eu tive foram causados por isso. A instalação foi possível só porque o próprio desenvolvedor me ajudou (obg Neal e obg Github).
Vocês podem baixar no site a versão que vocês quiserem - tanto a mais recente quanto a última estável. Vai vir como um tar.gz ou tar alguma coisa - é só um arquivo zippado. Unzip e abra um terminal dentro da pasta criada (ou troque o working directory com o cd).
O primeiro passo é criar o arquivo de configuração fazendo ./configure opções. Tem muitas opções. Pra quem tiver o gcc 10, você precisa colocar as seguintes coisas:
- CFLAGS=“-g -O3 -fcommon -ffp-contract=off”
- FFLAGS=“-fallow-argument-mismatch”
Portanto, o comando vai ser:
./configure CFLAGS="-g -O3 -fcommon -ffp-contract=off" FFLAGS="-fallow-argument-mismatch"Cuidado que espaço no lugar errado vai gerar um erro. Se você quer usar o pqR dentro do RStudio, você tem que adicionar também --enable-R-shlib. O comando fica:
./configure --enable-R-shlib CFLAGS="-g -O3 -fcommon -ffp-contract=off" FFLAGS="-fallow-argument-mismatch"Isso vai demorar um pouco e vai imprimir uma montanha de coisas no terminal. No fim, se tudo der certo, pode aparecer um warning ou outro. Um erro é um erro e você vai ter que fuçar a internet para ver se resolver - comece checando as coisas óbvias, como pacotes faltando e as instruções do pqR.
O passo seguinte é compilar o pacote, dando o comando make (sem necessidade de ./). Vai demorar um tempo e é totalmente normal aparecer warnings, coisas coloridas etc. Vá fazer um café ou leia outros posts do blog enquanto isso.
Se tudo der certo, ele vai sair sem dar nenhum erro. Eu achei um pouco obscuro quando deu erro, e no fim da compilação não aparece uma mensagem óbvia que foi bem sucedido, então leia com calma o que saiu no terminal.
Feito isso, você pode acessar o pqR executando, do terminal onde você instalou, ./bin/R. A mensagem de início do R deve te informar que você está usando o pqR. Veja que você pode começar a usar o pqR direto do terminal.
Se você quer usar no RStudio, esta página do RStudio explica. Basicamente você vai executar o comando export RSTUDIO_WHICH_R=pathtopqR no terminal. A parte importante: isso só vai alterar as seções do rstudio iniciadas do terminal, então você vai ter que iniciar o RStudio do terminal. O link comenta que esse é o padrão no Ubuntu e aparentemente também é o padrão no arch.
É possível trocar para o que é iniciado direto do icone da interface gráfica. Eu não tentei isso porque eu achei extremamente conveniente ter como iniciar o R “normal” e o pqR no RStudio sem precisar toda vez trocar um arquivo escondido em algum canto.
Se você acha meio inconveniente toda vez abrir o terminal, dá pra criar um icone na área de trabalho abrindo um arquivo em branco e salvando com o nome pqR.desktop e com o seguinte conteúdo:
#!/usr/bin/env xdg-open
[Desktop Entry]
Type=Application
Exec=sh -c 'export RSTUDIO_WHICH_R=pathto/pqR-2019-07-05/bin/R; rstudio-bin'
Icon=pathto/pqR-2019-07-05/doc/html/pqR.ico
Name=pqR
Terminal=true
GenericName=rstudio-term
Categories=Development;No ‘pathto’ troquem para o lugar onde vocês deram build do pqR. O GenericName e Categories não tem nada de especial .Veja que ele primeiro muda o caminho do R e depois executa o rstudio via terminal - aqui, quando eu fecho o terminal, ele restaura as variáveis do ambiente pro original. Eu não sei o quão universal é esse último comportamento entre as distros de Linux.
Eu não acho que o pqR vai substituir o R do CRAN tão cedo. Ele tem várias vantagens, especialmente se você quer mais velocidade. Ele tem desvantagens: primeiro, ele é baseado em uma versão antiga do R; segundo, o R é uma linguagem (de programação), e linguagens sós ervem se outras pessoas falam a mesma língua. Provavelmente projetos seus com co-autores vão depender do que tem no R usual, e você vai precisar do R base.