Classificando distribuições a partir dos momentos
Surgiu uma curiosidade legítima na minha cabeça ontem e eu queria saber se consigo, a partir dos momentos amostrais, treinar um classificador razoável para a família do processo gerador. Responder isso vai ser divertido porque é um bom playgrond para ferramentas do tidyverse e deixa de exemplo um fluxo mínimo de modelagem com tidymodels.
A primeira coisa a fazer é uma função que recebe um nome de função que possa gerar dados aleatórios seguindo algum processo conhecido - o parâmetro dgp vem de data generating process. Vou parametrizar ela menos do que é possível porque, putz moh trabalho.
process_factory <- function(dgp, n = 100) {
if(dgp == "rnorm") {
to_be_called <- call("rnorm",
n = n,
sd = runif(1, 0, 3),
mean = runif(1, -3, 3))
} else if(dgp == "rt") {
to_be_called <- call("rt",
n = n,
df = sample(1:30, 1),
ncp = runif(1, 0, 10))
} else if(dgp == "runif") {
to_be_called <- call("runif",
n = n,
min = runif(1, -3, 0),
max = runif(1, 0, 3))
} else if(dgp == "rexp") {
to_be_called <- call("rexp",
n = n,
rate = runif(1, 0, 3))
} else if(dgp == "rgamma") {
to_be_called <- call("rgamma",
n = n,
shape = runif(1, 0, 3),
scale = (1/runif(1, 0, 3)) + rnorm(1, sd = .2))
}
eval(to_be_called)
}Agora uma função que recebe um vetor com dados simulados com algum processo gerador dado e retorna os primeiros \(k\) momentos amostrais.
first_kmoments <- function(process, .min_k = 1, .k = 20, .center = TRUE) {
tibble(process = list(process)) %>%
list() %>%
rep(times = .k) %>%
reduce(bind_rows) %>%
mutate(K = .min_k:.k,
moment = map2_dbl(
.x = process,
.y = K,
~ moment(x = .x, order = .y, center = .center))) %>%
pivot_wider(values_from = moment,
names_from = K,
names_prefix = "moment_") %>%
select(-process)
}(Tem um bug bem fácil de consertar e de reproduzir na função acima, fica como exercício)
Beleza agora podemos de fato simular alguns dados e pedir os momentos das distribuições simuladas.
library(tidyverse)
library(e1071)
library(magrittr)
(tibble(
dgp = # variável com o nome dos processos, o Y do nosso modelo
sample(c("rnorm", "runif", "rexp", "rgamma", "rt"), # amostre um de 4 nomes
size = 10000, # 100000 vezes
replace = TRUE), # com substituição
moments = # variável momentos que irá conter uma lista de dataframes
map(dgp, # itere sobre o vetor com nomes de dgps
~ first_kmoments(process_factory(.x)))) %>%
unnest(moments) %>% # desaninha a lista de dataframes
na.omit() ->
data)## # A tibble: 9,981 x 21
## dgp moment_1 moment_2 moment_3 moment_4 moment_5 moment_6 moment_7
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 runif 2.22e-18 2.47 -8.95e-3 1.02e+1 3.48e-2 4.91e+ 1 1.49e+ 0
## 2 rt 3.20e-16 12.4 6.30e+1 9.49e+2 1.11e+4 1.57e+ 5 2.23e+ 6
## 3 runif -1.11e-17 0.528 -3.29e-2 5.86e-1 -1.25e-1 8.24e- 1 -3.28e- 1
## 4 rnorm 2.22e-16 0.970 2.19e-1 2.20e+0 1.14e+0 6.63e+ 0 5.35e+ 0
## 5 rt 5.86e-16 2.59 2.10e+0 2.10e+1 4.57e+1 2.86e+ 2 9.57e+ 2
## 6 rt 6.39e-16 826. 1.20e+5 2.07e+7 3.68e+9 6.62e+11 1.20e+14
## 7 runif -2.50e-18 0.0904 8.14e-4 1.45e-2 -8.97e-5 2.81e- 3 -1.03e- 4
## 8 runif 1.61e-17 0.457 -4.20e-2 3.73e-1 -7.06e-2 3.72e- 1 -1.04e- 1
## 9 runif 1.55e-17 0.831 -1.32e-1 1.23e+0 -4.43e-1 2.18e+ 0 -1.20e+ 0
## 10 rt -2.13e-16 2.47 3.75e+0 2.51e+1 8.53e+1 4.46e+ 2 1.97e+ 3
## # … with 9,971 more rows, and 13 more variables: moment_8 <dbl>,
## # moment_9 <dbl>, moment_10 <dbl>, moment_11 <dbl>, moment_12 <dbl>,
## # moment_13 <dbl>, moment_14 <dbl>, moment_15 <dbl>, moment_16 <dbl>,
## # moment_17 <dbl>, moment_18 <dbl>, moment_19 <dbl>, moment_20 <dbl>Como são dados 100% simulados eu não vejo a virtude de explorar graficamente, vou pular então. Agora vamos treinar uma grade de modelos Random Forest.
library(tidymodels)
doParallel::registerDoParallel() # executar em paralelo
data_split <- initial_split(data, strata = dgp) # divisão dos dados
data_treino <- training(data_split) # treino
data_teste <- testing(data_split) # teste
data_rec <- recipe(dgp ~ ., data = data_treino)
data_prep <- prep(data_rec)
(modelo_tune <- rand_forest( # especificação da grade a ser avaliada
mtry = tune(),
trees = 1000,
min_n = tune()) %>%
set_mode("classification") %>%
set_engine("randomForest"))## Random Forest Model Specification (classification)
##
## Main Arguments:
## mtry = tune()
## trees = 1000
## min_n = tune()
##
## Computational engine: randomForest(modelo_workflow <- workflow() %>%
add_recipe(data_rec) %>%
add_model(modelo_tune))## ══ Workflow ═══════════════════════════════════════════════════════════════════════════════════════════════
## Preprocessor: Recipe
## Model: rand_forest()
##
## ── Preprocessor ───────────────────────────────────────────────────────────────────────────────────────────
## 0 Recipe Steps
##
## ── Model ──────────────────────────────────────────────────────────────────────────────────────────────────
## Random Forest Model Specification (classification)
##
## Main Arguments:
## mtry = tune()
## trees = 1000
## min_n = tune()
##
## Computational engine: randomForest(data_folds <- vfold_cv(data_treino, 5))## # 5-fold cross-validation
## # A tibble: 5 x 2
## splits id
## <list> <chr>
## 1 <split [6K/1.5K]> Fold1
## 2 <split [6K/1.5K]> Fold2
## 3 <split [6K/1.5K]> Fold3
## 4 <split [6K/1.5K]> Fold4
## 5 <split [6K/1.5K]> Fold5O passo final é “afinar” a grade e estimar todos os modelos seguindo o workflow.
(tune_results <- tune_grid(
modelo_workflow,
resamples = data_folds,
grid = 20
))Vamos avaliar brevemente a área abaixo da curva ROC dos vários modelos que treinamos.
tune_results %>%
collect_metrics(summarize = FALSE) %>%
filter(.metric == "roc_auc") %>%
ggplot(aes(x = .estimate)) +
geom_histogram(fill = "light green") +
labs(title = "Distribuição das AUROC",
x = "AUROC",
y = "") +
scale_x_continuous(labels = scales::percent) +
theme_minimal()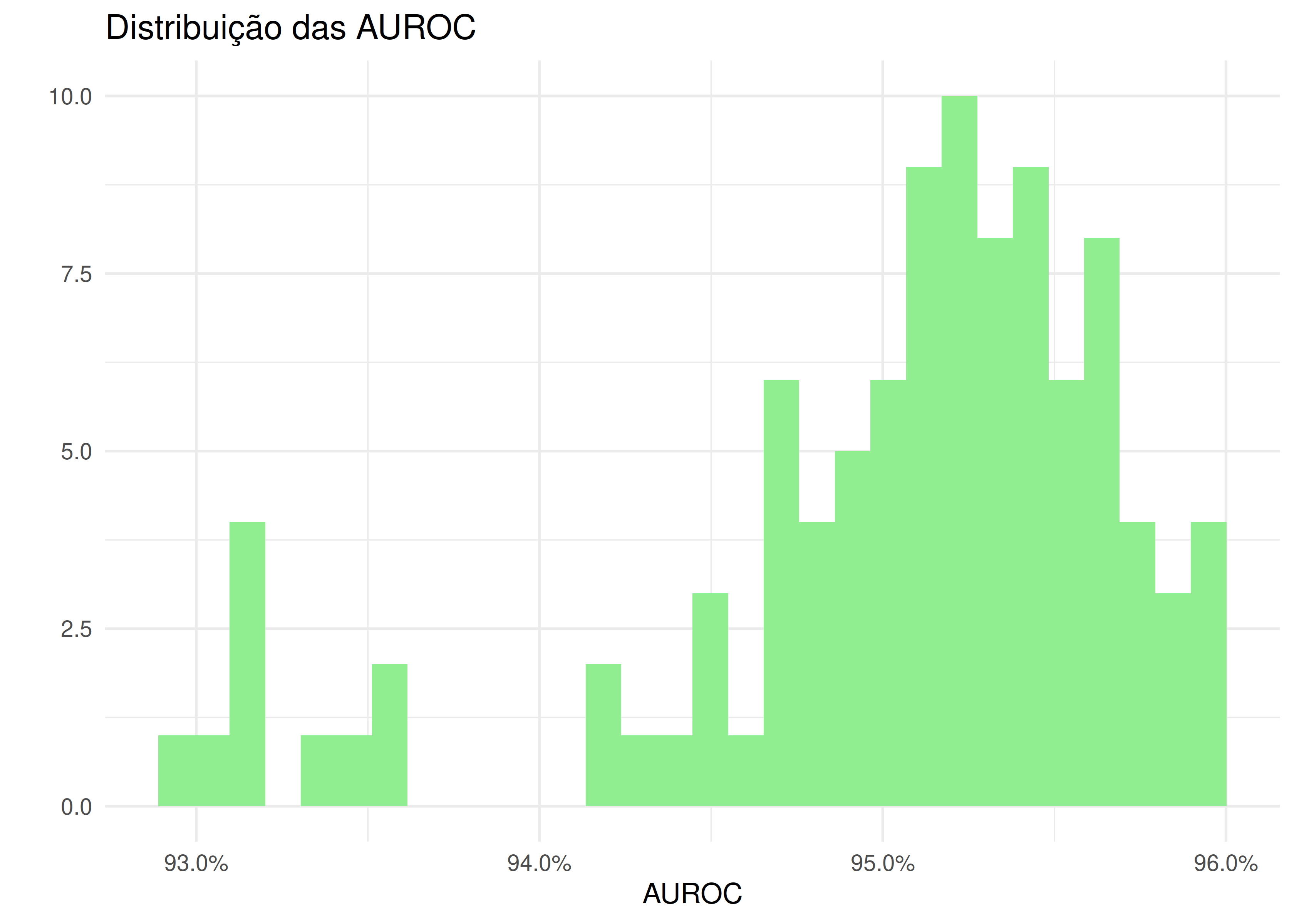
A primeira vez que rodei tudo me deu AUROCs beeem altas, além dos 95%. Em classificação binária, na maioria dos domínios de aplicação, isso é alto demais para ser verdade, mas problemas multiclasse normalmente vêm com AUROCs altas. Vamos olhar com mais cuidado para o melhor modelo - ainda dentro da amostra de treino.
library(vip)
(melhor_auc <- select_best(tune_results, "roc_auc"))## # A tibble: 1 x 3
## mtry min_n .config
## <int> <int> <chr>
## 1 14 9 Model01(modelo_final <- finalize_model(modelo_tune, melhor_auc))## Random Forest Model Specification (classification)
##
## Main Arguments:
## mtry = 14
## trees = 1000
## min_n = 9
##
## Computational engine: randomForest(avaliacao <- modelo_final %>%
set_engine("randomForest") %>%
fit(dgp ~ ., data = juice(data_prep)))## parsnip model object
##
## Fit time: 16.4s
##
## Call:
## randomForest(x = as.data.frame(x), y = y, ntree = ~1000, mtry = ~14L, nodesize = ~9L)
## Type of random forest: classification
## Number of trees: 1000
## No. of variables tried at each split: 14
##
## OOB estimate of error rate: 21.09%
## Confusion matrix:
## rexp rgamma rnorm rt runif class.error
## rexp 1024 387 0 48 0 0.29814942
## rgamma 514 768 8 140 1 0.46331237
## rnorm 0 12 1309 160 37 0.13768116
## rt 40 93 110 1320 6 0.15869981
## runif 0 6 16 1 1488 0.01522171Interessante que algumas distribuições são particularmente difíceis de acertar, como a gama. A uniforme e a normal, por exemplo, são beeem mais fáceis de acertar.
vip(avaliacao, geom = "col", fill = "light green") +
labs(title = "Importância de variáveis para o classificador") +
theme_minimal()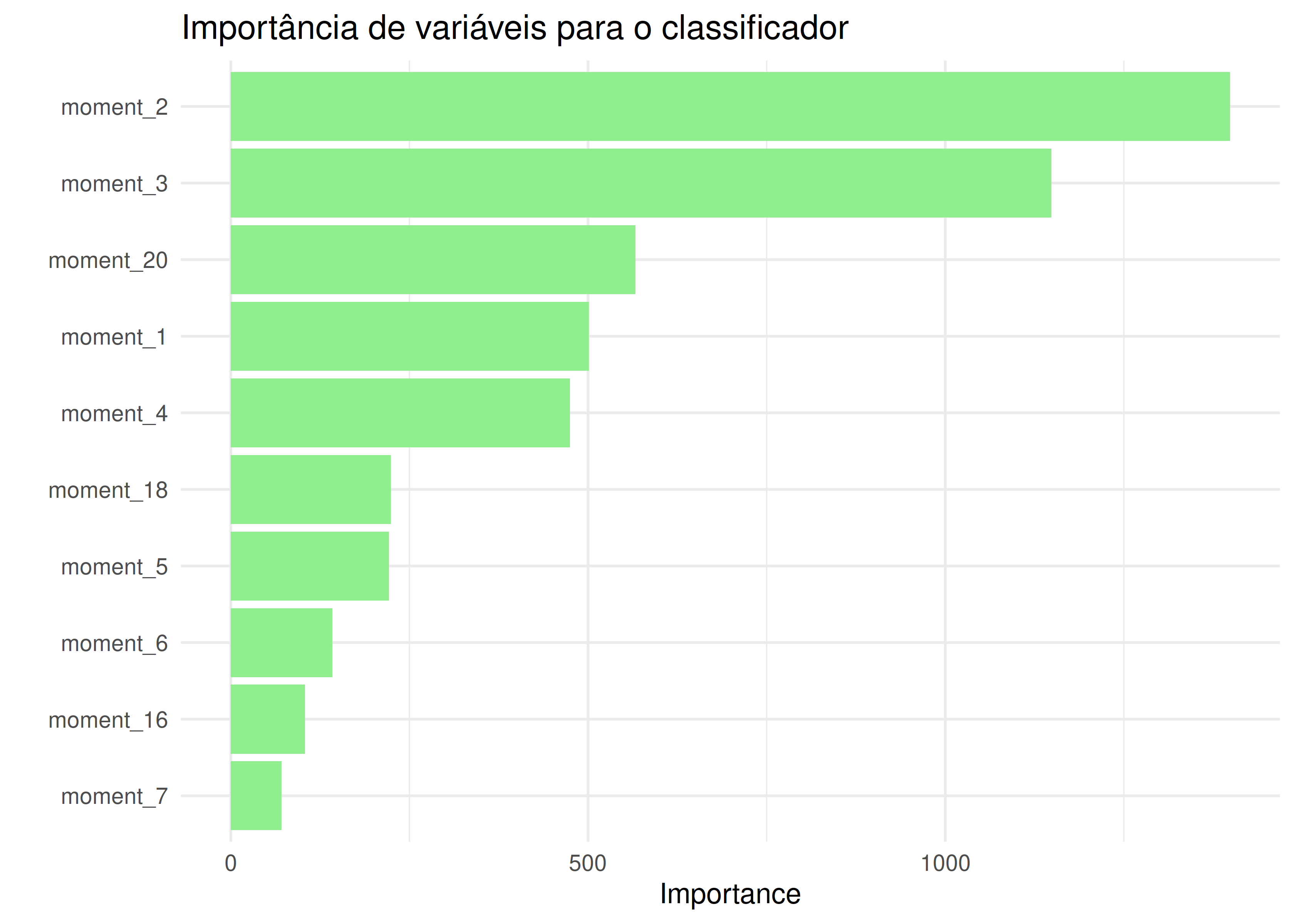
Acho razoável pensar que os momentos mais discriminantes dependem da cesta de distribuições alimentadas. É difícil pensar que assimetria é relevante para distinguir entre uma normal e uma uniforme, por exemplo.
E como ficamos fora da amostra de teste? A função last_fit() pega a receita de dados de treino e aplica na amostra de teste.
workflow_final <- workflow() %>%
add_recipe(data_rec) %>%
add_model(modelo_final)
(final_res <- workflow_final %>%
last_fit(data_split))## # Resampling results
## # Monte Carlo cross-validation (0.75/0.25) with 1 resamples
## # A tibble: 1 x 6
## splits id .metrics .notes .predictions .workflow
## <list> <chr> <list> <list> <list> <list>
## 1 <split [7.5K… train/test… <tibble [2 ×… <tibble [0… <tibble [2,493 … <workflo…final_res %>%
collect_metrics()## # A tibble: 2 x 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy multiclass 0.791
## 2 roc_auc hand_till 0.957A pergunta era se dá. Até que dá.